딥러닝 기반 CT 스캔 재구성을 통한 조영제 사용 및 신체 부위 분류 성능 향상 연구
Copyright © 2023, The Korean Institute of Broadcast and Media Engineers
This is an Open-Access article distributed under the terms of the Creative Commons BY-NC-ND (http://creativecommons.org/licenses/by-nc-nd/3.0) which permits unrestricted non-commercial use, distribution, and reproduction in any medium, provided the original work is properly cited and not altered.
초록
표준화되지 않은 의료 데이터 수집 및 관리는 여전히 수동으로 진행되고 있어, 이 문제를 해결하기 위해 딥 러닝을 사용해 CT 데이터를 분류하는 연구들이 진행되고 있다. 하지만 대부분 연구에서는 기본적인 CT slice인 axial 평면만을 기반으로 모델을 개발하고 있다. CT 영상은 일반 이미지와 다르게 인체 구조만 묘사하기 때문에 CT scan을 재구성하는 것만으로도 더 풍부한 신체적 특징을 나타낼 수 있다. 이 연구는 axial 평면뿐만 아니라 CT 데이터를 2D로 변환하는 여러가지 방법들을 통해 보다 높은 성능을 달성할 수 있는 방법을 찾고자 한다. 훈련은 5가지 부위의 CT 스캔 1042개를 사용했고, 모델 평가를 위해 테스트셋 179개, 외부 데이터셋으로 448개를 수집했다. 딥러닝 모델 개발을 위해 ImageNet으로 사전 학습된 InceptionResNetV2를 백본으로 사용하였으며, 모델의 전체 레이어를 재 학습했다. 실험결과 신체 부위 분류에서는 재구성 데이터 모델이 99.33%를 달성하며 axial 모델보다 1.12% 더 높았고, 조영제 분류에서는 brain과 neck에서만 axial모델이 높았다. 결론적으로 axial slice로만 훈련했을 때 보다 해부학적 특징이 잘 나타나는 데이터로 학습했을 때 더 정확한 성능 달성이 가능했다.
Abstract
Unstandardized medical data collection and management are still being conducted manually, and studies are being conducted to classify CT data using deep learning to solve this problem. However, most studies are developing models based only on the axial plane, which is a basic CT slice. Because CT images depict only human structures unlike general images, reconstructing CT scans alone can provide richer physical features. This study seeks to find ways to achieve higher performance through various methods of converting CT scan to 2D as well as axial planes. The training used 1042 CT scans from five body parts and collected 179 test sets and 448 with external datasets for model evaluation. To develop a deep learning model, we used InceptionResNetV2 pre-trained with ImageNet as a backbone and re-trained the entire layer of the model. As a result of the experiment, the reconstruction data model achieved 99.33% in body part classification, 1.12% higher than the axial model, and the axial model was higher only in brain and neck in contrast classification. In conclusion, it was possible to achieve more accurate performance when learning with data that shows better anatomical features than when trained with axial slice alone.
Keywords:
artificial intelligence, computed tomography, deep learning, transfer learning, two-dimensional convert methodI. 서 론
최근 Deep Neural Network은 컴퓨터 비전 분야에서 기존 방법들보다 높은 성능을 달성하고 있다. 컴퓨터 비전 분야 중 하나인 분류 문제는 이미 인간보다 높은 정확도를 달성하였다[1]. 이러한 DLM(Deep Learning Model)의 강력한 성능으로 인해 지난 몇 년간 AI기술을 도입하여 의료 영상 응용프로그램 및 자동 파이프 라인을 개발하는 것에 대한 관심이 증가하였다[2]. 그러나 개발된 응용프로그램의 적용은 병원 ecosystem, 방사선 플랫폼, 프로세스 및 프로토콜의 이질성을 비롯한 여러 가지 이유로 어렵다[3]. 또한 데이터 획득 시 수동 개입이 포함될 수 있으며, 동일한 기관에서는 스캐너 제조업체, 모델 및 소 프트웨어 버전에 상당한 이질성이 있으며 이러한 변동성으로 인해 획득한 파라미터와 관련 메타데이터들은 환자마다 다를 수 있다[4].
이런 문제들로 인해 의료 데이터들은 표준화되기 어렵고, 데이터 관리 및 연구 데이터 수집을 위해서는 영상 픽셀의 특성에 의존하여 영상을 식별하는 방사선 전문가가 수동으로 처리해야 하며 모든 데이터를 검수하는 것은 병원 측면에서 상당한 비용이 필요하다. 위와 같은 문제들을 해결하기 위해 이전 일부 연구에서는 딥러닝[5,6]을 사용하여 CT(Computed Tomography)영상을 분류했지만 일정한 CT slice 위치에서만 분류 정확도를 평가했다. 방사선 전문가들은 일반적으로 slice 위치, 장기 구조 상태 및 종양 강화 존재를 참조하기 때문에, 종양 진단을 위해 조영제를 사용하는 증강 정보의 필요성 또한 인식되고 있다[7,8]. 하지만 현재까지 조영 증강 데이터를 포함하여 CT 영상 분류에 관한 연구는 많지 않다. 이와 관련된 연구로 조영제와 신체 부위 분류를 수행한 논문[9]이 보고되었지만 모델의 정확성과 분류 모델을 만들기 위한 CT 이미지의 수에 대해 설명하였다. 이 논문에서는 axial 영상을 0.1K에서 10K까지 사용하면서 실험을 진행했지만 임상에서 사용할 수 있는 수준의 정확도 (accuracy, 72% ~ 86%)를 달성하진 못했다. 그리고 복부 slice의 조영 증강 분류에만 초점을 맞춘 연구[10]도 있었지만 이 논문은 너무 일부에만 초점을 맞췄음에도 정확도가 높지 않다. 대부분의 CT 데이터 기반의 분류과제 논문들은 모두 기본적인 CT slice(axial plane)를 사용하여 AI모델들을 학습하였다. 하지만 CT 영상은 일반 이미지와 다르게 인체 이외의 배경이 없고 인체 구조만 묘사하기 때문에 3D로 구성하고 신체 해부학적으로 판단하기 쉽게 2D로 재구성한다면 axial 영상보다 더 풍부한 신체적 특징을 학습할 수 있을 것이다. 이 논문에서는 기존 axial 영상뿐만 아니라 CT Scan을 3D로 재구성하고 2D 영상으로 변환하는 방법들을 사용하여 각각의 데이터 셋을 구축해 DLM을 학습하고, 기존 방법보다 신체 부위와 조영제 사용 분류 성능을 높일 수 있는 방법들이 있는지 실험을 통해 분석해보고자 한다.
II. 데이터 셋
1. Data source and Datasets
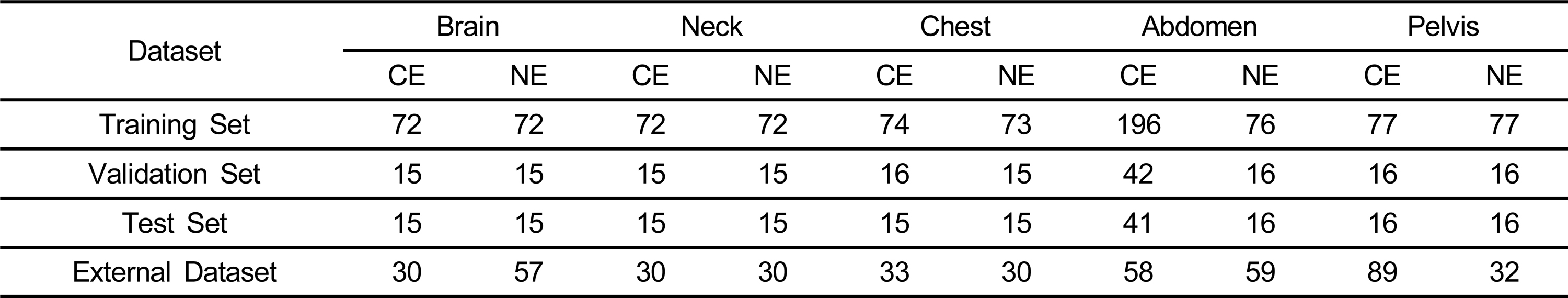
이 연구는 조영제 사용 및 신체 부위를 분류하기 위해 Brain, Neck, Chest, Abdomen, Pelvis 5가지 부위의 CT 스캔을 사용하였다. 먼저 모델 훈련에 사용된 데이터는 1042개로 Training Set의 경우 861개, Validation Set은 181개로 구성되었으며 이는 무작위로 선택되었다. 모델의 성능을 정확하게 측정하기 위한 Test Set과 External Dataset은 179개와 448개로 각각 구성되었으며, Contrast와 Non-contrast의 비율을 1:1로 맞추고자 노력하였다. Test Set과 External Dataset은 개발된 모델의 독립적인 성능 테스트에만 사용되고 훈련에는 사용되지 않았다. 표 1은 각 데이터 셋 별 데이터 수를 나타낸다.
III. CT Scan 3D 재구성 방법
1. CT Data
CT는 X-선을 신체에 투과, 그 흡수차이를 컴퓨터로 재구성하여 인체의 단면 영상(Cross-sectional Image)을 얻거나 3차원적인 입체 영상을 얻는 영상 진단 법이다. CT 기기에서 나선형 스캔을 통해 체적 데이터를 획득 가능하며 이를 3D로 재구성할 수 있다. 그림 1은 CT slice의 단면과 단면들을 결합하여 3D로 재구성한 예를 보여준다.
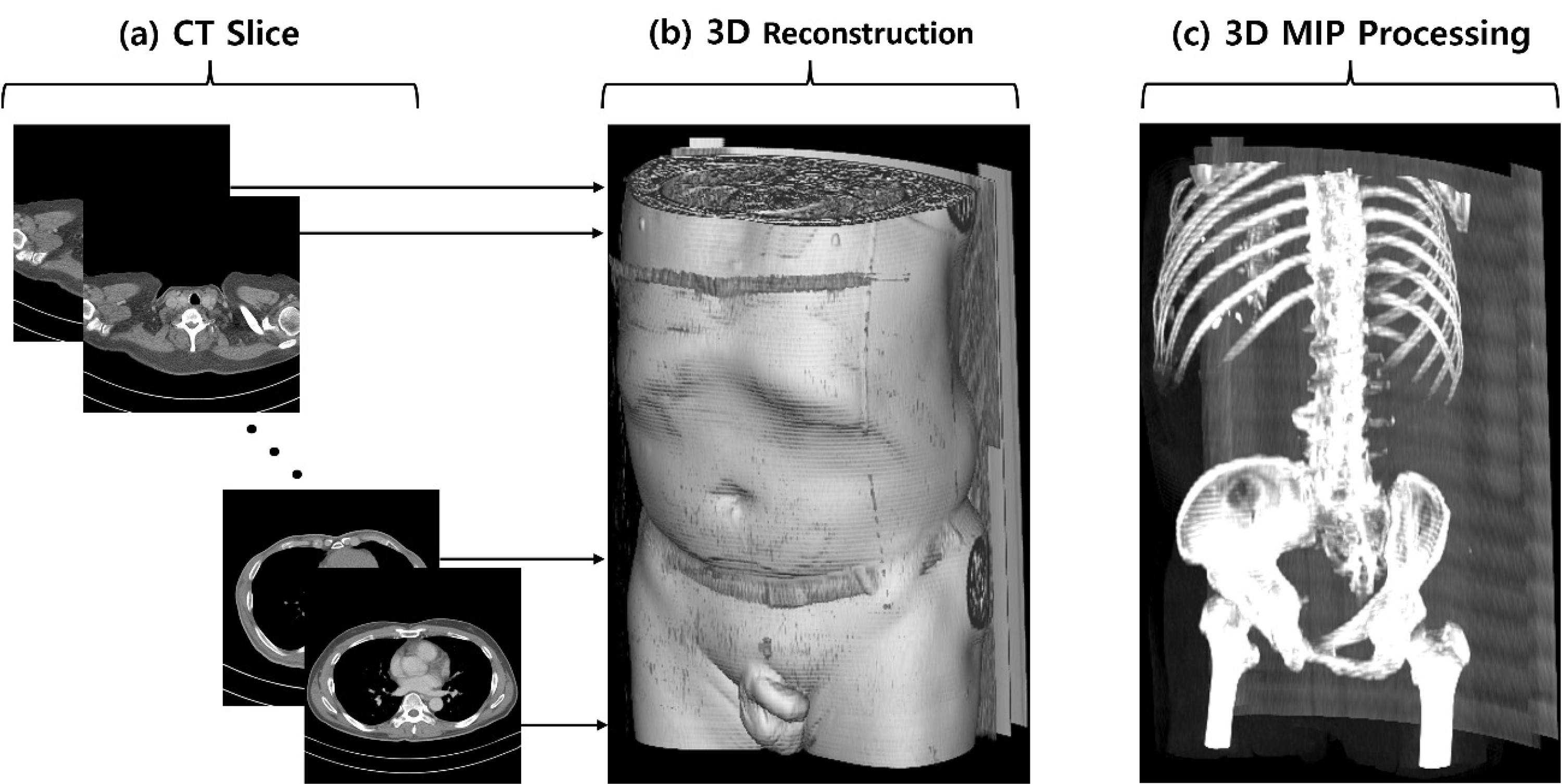
CT Slice와 3D 재구성 방법. (a) CT 기기에서 획득된 CT slice 단면들, (b) CT slice 단면들을 결합하여 3D로 재구성한 예, (c) CT slice를 MIP(Maximum Intensity Projection) 방법으로 재구성한 예Fig. 1. CT Slice and 3D reconstruction methods. (a) CT slice obtained from a CT device, (b) Example of 3D reconstruction by combine CT slice (c) Example of a MIP (Maximum Intensity Projection) reconstruction of a CT slice.
2. CT Scan 재구성 방법
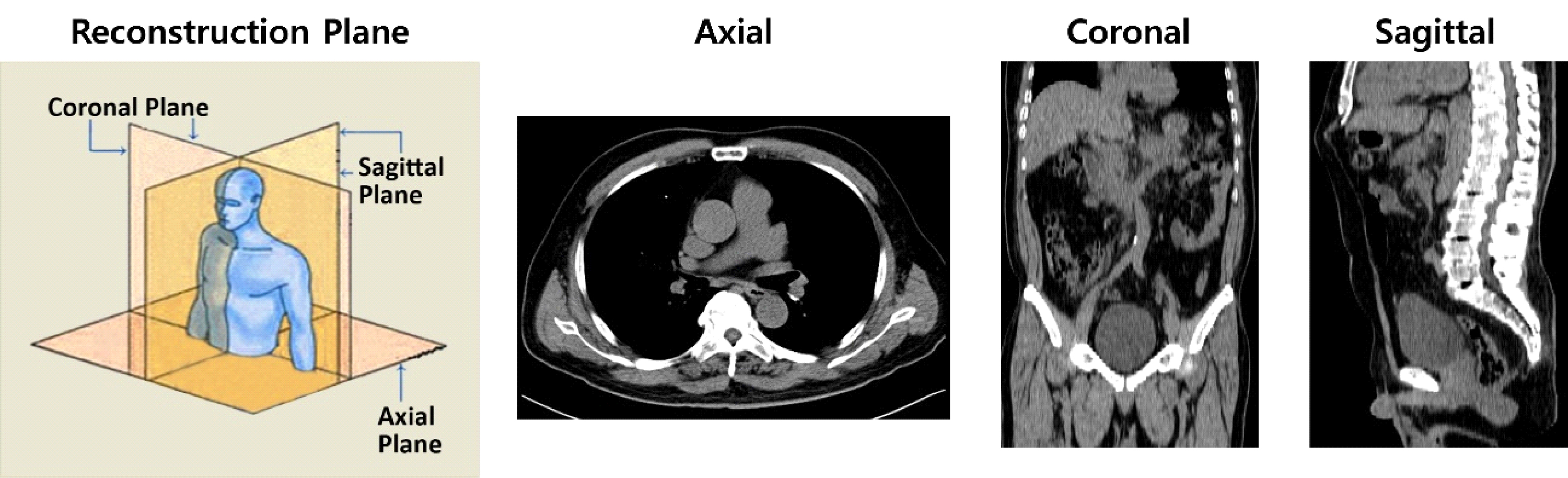
CT Scan는 여러가지 방법들로 재구성할 수 있다. 3D CT 데이터를 2D로 변환 시 X, Y, Z 축을 기반으로 2D 평면에 투영할 수 있으며, 그림 2에서와 같이 X축으로 투영하면 시상면(Sagittal), Y축은 수평면(Axial), Z축이 기반이면 관상면(Coronal)으로 재구성할 수 있다. 이렇게 변환된 2D 영상은 판독 시 각각 이점이 있기 때문에 영상의학에서 많이 사용되는 전처리 방법이다. 또한 2D 평면으로 투영 시 픽셀 값 또한 3가지 방법으로 재구성할 수 있다. 그림 1의 MIP Processing에서와 같이 2D 평면으로 3D 데이터를 투영 시 해당 픽셀에서 가장 높은 값으로만 재구성하는 것을 MIP 방법이라고 하며, 또 다른 방법으로는 AIP와 Mid-plane이 있다. AIP(Average Intensity Projection)는 2D평면으로 투영 시 모든 slice들에서 해당 픽셀 값의 평균으로 2D 평면 픽셀 값을 재구성한다. Mid-plane은 각 축 별 CT slice 중 가운데 slice를 선택한다.
IV. Deep Learning Model 개발
1. 재구성 데이터 별 DLM Training
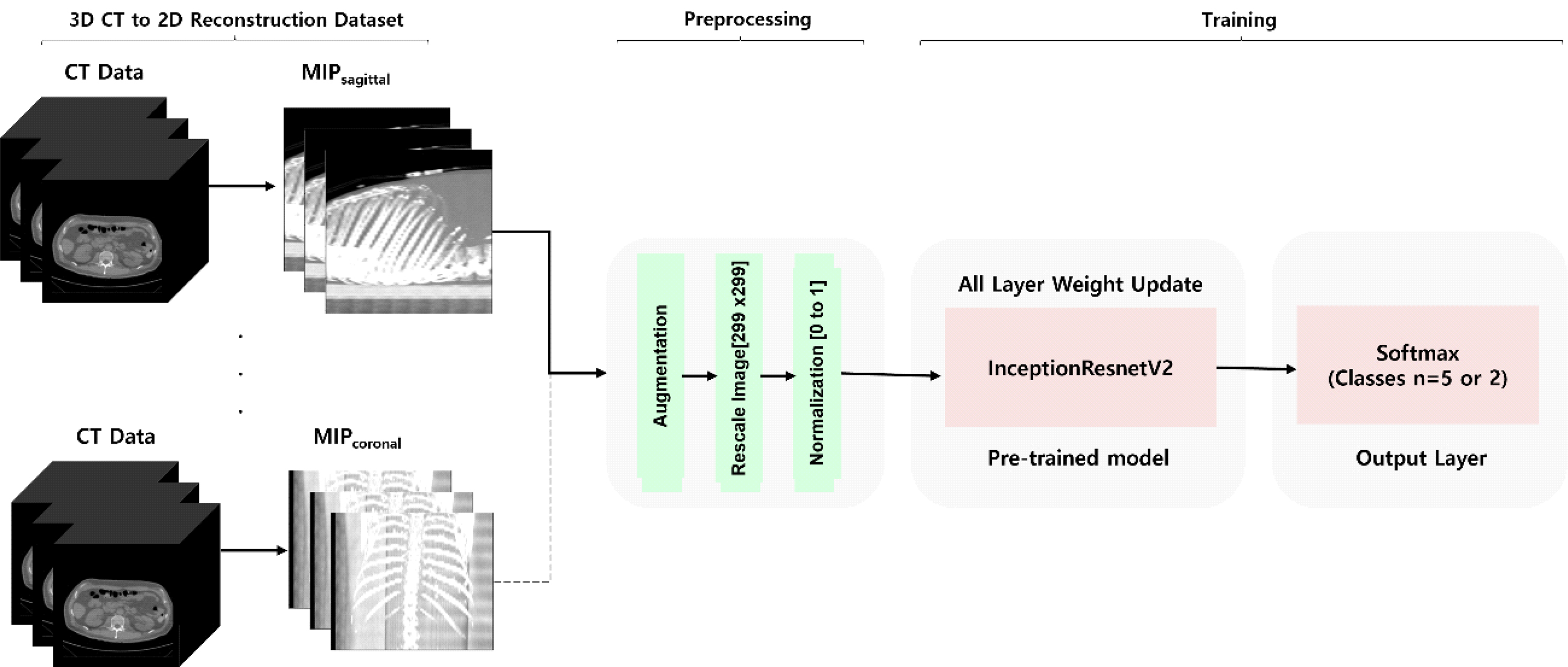
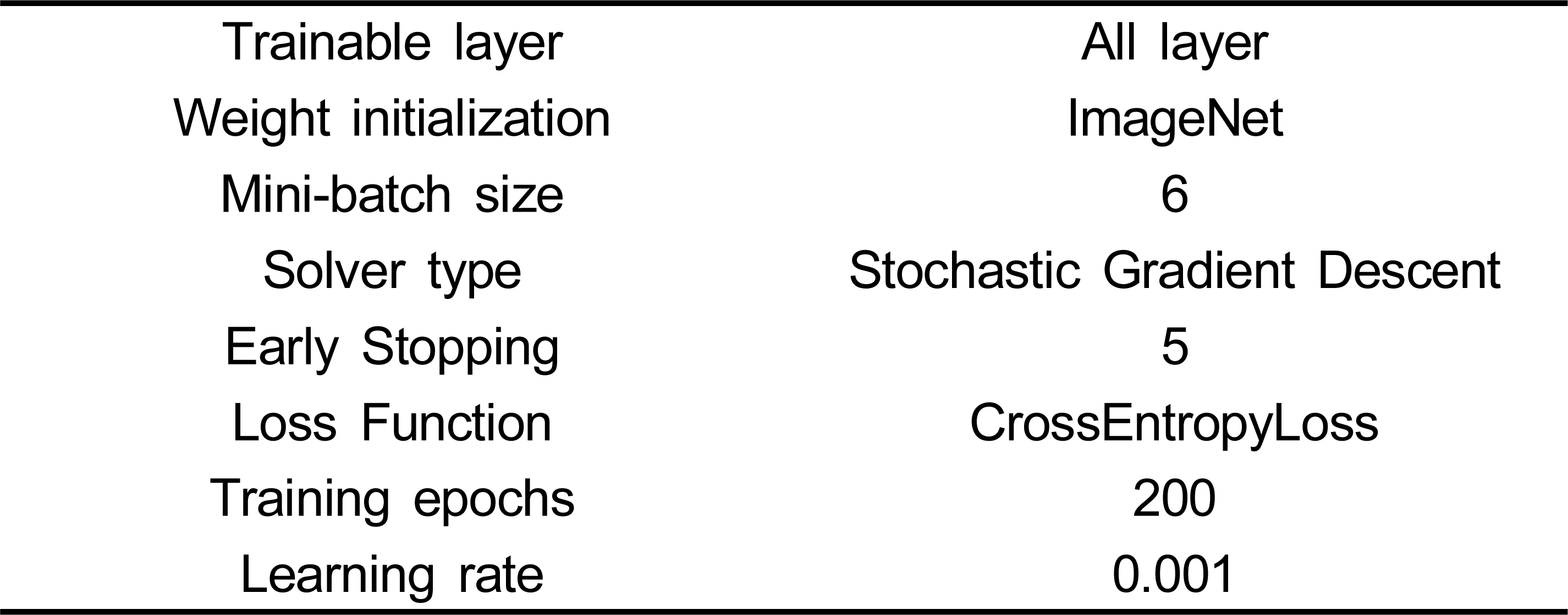
이 연구에서는 조영제 사용과 신체 부위 분류를 위한 모델을 각각 개발하였으며, 위에서 설명한 CT Scan 데이터를 재구성하는 9가지 방법들(AIP, Mid-plane, MIPaxial, coronal, sagittal)을 사용하여 구성된 각각의 다른 데이터셋들을 통해 각각의 개별 DLM(Deep Learning Model)들을 훈련하였다. DLM은 ImageNet데이터로 사전 학습된 InceptionRexnetV2[11]를 백본으로 각각의 데이터 셋들을 사용하여 Transfer learning을 통해 개발하였다. 각각의 DLM은 표 2와 같은 Hyperparameter로 설정되어 학습되었다.
그림 3은 DML 훈련 개요를 보여준다. 먼저 3D CT 데이터를 2D로 재구성하는 방법들을 통해 얻어진 각각의 데이터 셋을 사용하여 개별 모델들을 훈련한다. 훈련 데이터 셋은 백본 네트워크에 입력 전 전처리 단계를 먼저 통과한다. 전처리 단계에서는 Augmentation, Rescale, Normalization이 수행된다. Augmentation은 Random Flip, Rotate, Zooming, Gaussian noise, Wrapping의 기법들을 사용하여 수행되었다. 그 후 영상의 width, height를 299로 Rescale하고, 픽셀 값들을 0~1사이로 만들어 네트워크 입력값을 정규화 해준다. 전처리 단계 이후에는 InceptionResnetV2 네트워크에 입력하여 훈련을 진행하며, 이 때 모든 계층의 가중치를 업데이트 한다. 이러한 선택은 반복된 실험 결과에 따라 결정되었으며, 출력 계층은 Flatten과 Softmax 계층으로 구성되었다.
2. 모델 평가
Test Set 및 External Dataset을 사용하여 기본 CT slice 데이터인 axial평면 DLM과 (AIPaxial, Mid-planeaxial, MIPaxial)과 이 논문에서 제안하는 여러가지 재구성 데이터를 통해 훈련된 DLM들(AIPcornal,sagittal, Mid-planecornal,sagittal, MIPcornal,sagittal)의 성능을 평가했다. 성능 정확도 평가를 위해 Accuracy, precision, recall 가 사용되었다. Accuracy, precision, recall는 다음과 같이 정의된다.
여기서 TP, TN, FP, FN은 각각 True Positive, True Negative, False Positive, False Negative를 나타낸다.
V. 결과
1. 신체 부위 분류 성능
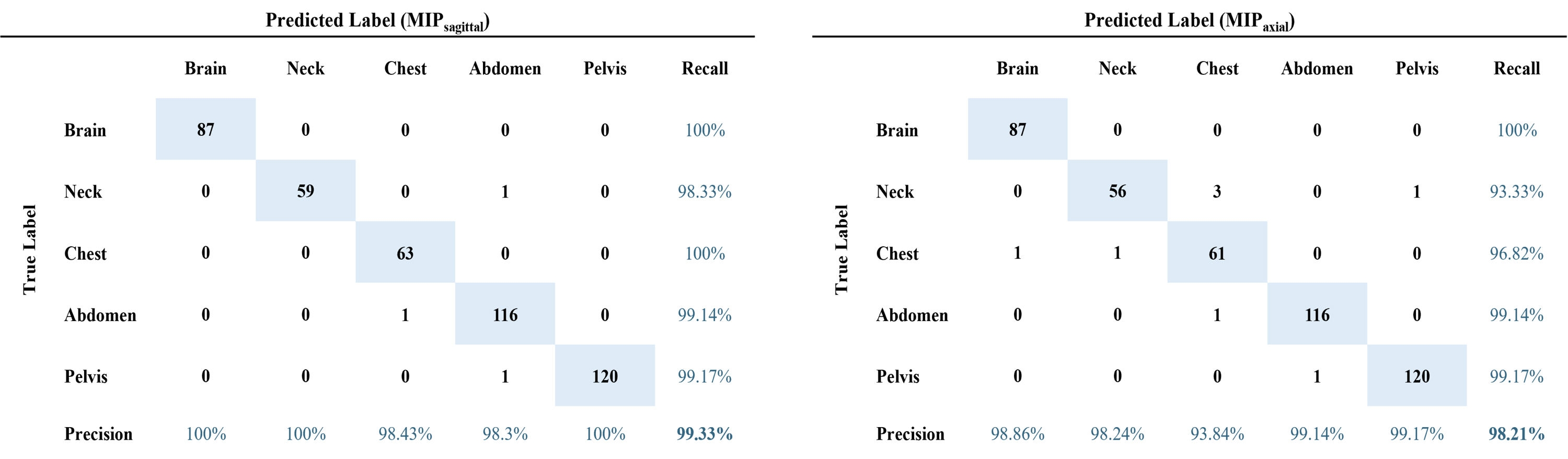
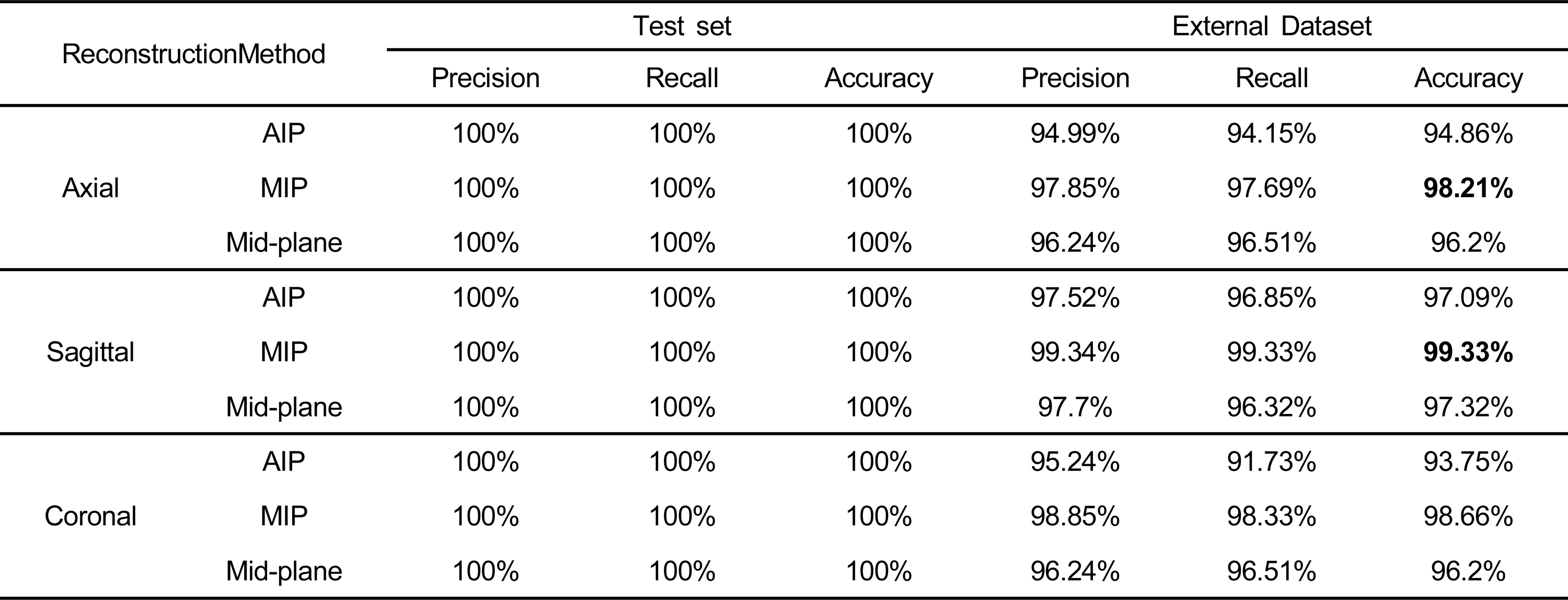
표 3은 재구성 데이터 별 DLM의 신체 부위 분류 성능을 보여준다. Test Set에서는 표 3에서 제시된 바와 같이 모든 DLM 성능(Accuracy, 100%)이 우수했다. External Dataset에서는 Sagittal 평면이 다른 평면들의 DLM보다 모든 픽셀 재구성 방법(AIP, MIP, Mid-plane)에서 높은 성능(Accuracy, 97.09%, 99.33%, 97.32%)을 보여준다. 그리고 모든 평면에서 MIP 방법의 픽셀 구성 DLM 성능은 다른 방법들에 비해 높은 성능을 보여준다.

재구성 데이터별 DLM의 신체 부위 분류 성능Table 3. Performance of body part classification of DLM by reconstruction data
External Dataset로 평가한 Axial 평면의 최고 성능 DLM(MIPaxial)과 재구성 데이터 별 DLM중 최고 성능 모델(MIPsagittal)의 혼동 매트릭스는 그림 4와 같다. External Dataset에서 측정된 최고 성능 DLM(MIPsagittal)은 3개의 case에서 불일치하였으며, MIPaxial DLM의 경우는 8개의 case에서 불일치 결과를 보였다.
2. 조영제 사용 분류 성능
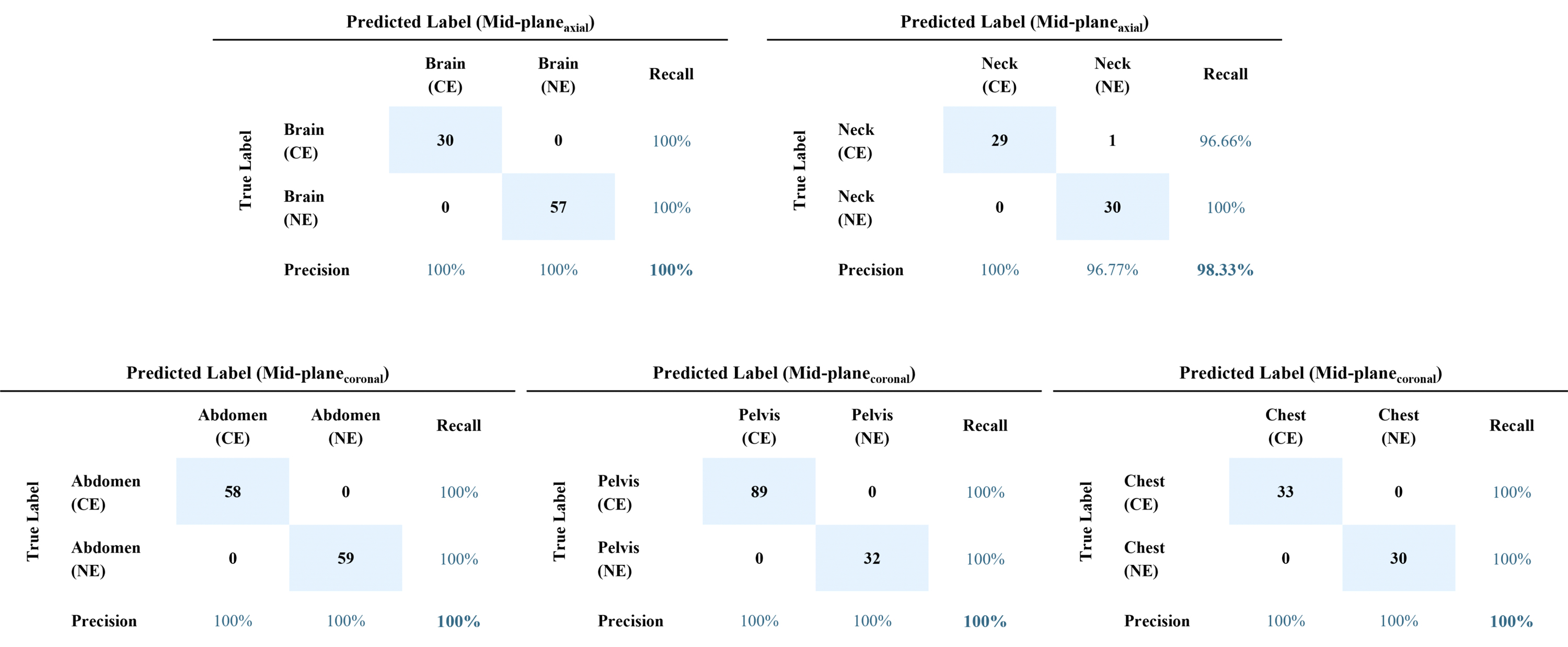
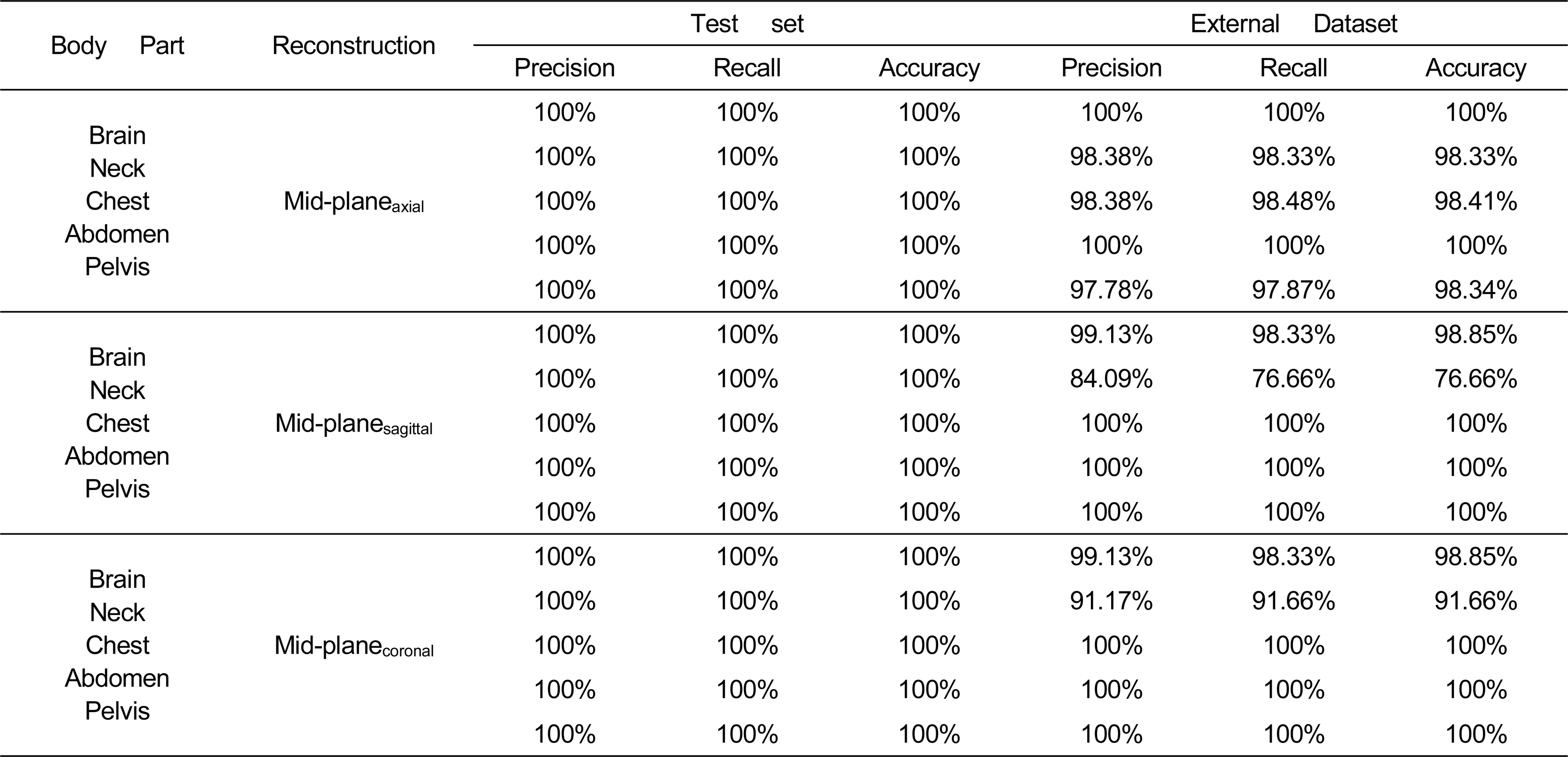
표 4는 재구성 데이터 별 DLM의 조영제 사용 분류 성능을 보여준다. Test Set에서 Mid-plane 기반의 DLM(accuracy, 94~100%)은 모든 신체 부위에서 MIP 또는 AIP 기반 DLM(accuracy, <90%)에 비해 높은 성능을 보였다. 신체부위 분류와는 다르게 Axial 평면 기반의 DLM은 Brain과 Neck 부위에서 조영제 사용 분류 성능이 다른 평면 기반 DLM들보다 높았다. 그 외 부위에서는 Sagittal과 Coronal 평면에서 더 좋은 성능을 보여줬다.

Mid-plane기반 DLM들의 신체 부위 별 조영제 사용 여부 분류 성능Table 4. Performance of contrast enhancement classification of mid-plane based DLMs
그림 5는 각 부위에서 조영제 사용 분류 성능이 가장 높은 DLM의 혼돈 행렬을 보여준다.
VI. 논의
이 연구는 기본 CT slice(axial) 영상을 기반으로 학습했던 DLM보다 CT Scan을 3D로 재구성하고, 재구성된 데이터 셋을 기반으로 학습한 DLM이 더 정확할 수 있음을 보여주었다. 신체 부위 분류에서 Axial 기반 DLM은 MIPaxial이 External Dataset에서 98.21% 정확도를 달성하며 가장 높은 성능을 보였지만, 최고 성능을 달성한 MIPsagittal DLM성능(accuracy, 99.33%)보다 낮았다. 이러한 성능 차이는 선택된 평면에 기인될 수 있으며, axial 평면에서는 Chest와 Neck 또는 Abdomen과 Pelvis가 유사하게 보일 수 있기 때문에 다른 평면에서 보다 더 많은 불일치 케이스가 발생한다. 하지만 조영제 사용 분류에서는 Mid-planeaxial이 Brain과 Neck에서 가장 높은 성능(accuracy, 100%, 98.33%)을 달성했다. 이는 sagittal과 coronal 평면에 조영제 증강 분류의 정확성을 저해하는 강력한 강화 기관이 포함되어 있을 수 있기 때문이다.
지금까지 CT Scan을 여러가지 방법으로 재구성하여 DLM을 개발한 연구는 거의 없다. Philbrick et al.은 CT Scan에서 복부 위치의 axial slice에 대해 조영제 사용을 예측하는 CNN을 개발했지만 복부 이외에는 사용할 수 없기 때문에 수동으로 복부 Slice를 선택해야 되며, 좁은 범위 밖에 적용하지 못한다. Sugimori et al.은 다양한 CNN 아키텍처를 사용하여 조영제 사용과 신체 부위 분류를 동시에 수행하는 Deep Learning Network를 개발했다. 하지만 모델의 정확도가 우리 연구 정확도(accuracy, 99.3%) 보다 낮은 72~86%로 측정됐다.
VII. 결론
이 논문에서는 기존 CT 분류 시 사용되었던 axial 평면 데이터 기반 DLM의 해부학적 단점을 보완하고자 3D CT Scan 데이터를 하나의 2D 이미지로 재구성하는 방법들 AIP, MIP, Mid-planeaxial,coronal,sagittal을 사용하여 각각의 데이터 셋을 구성하고, 이를 기반으로 개별 DLM 들을 학습했다. 각각의 DLM의 성능 평가를 위해 훈련에 사용되지 않은 Test Set과 타 기관 데이터인 External Dataset을 구성하여 일반화 성능도 같이 측정하였다. 결과적으로 신체 부위 분류에서는 axial 평면보다 sagittal 평면에서 더 정확한 성능을 달성하였으며, 조영제 사용 분류에서는 axial 평면이 brain과 neck에서 가장 높은 성능을 달성하였지만 chest, abdomen, pelvis에서는 sagittal과 coronal 평면에서 가장 높은 성능을 달성하였다. 연구 결과에서 알 수 있듯 axial slice로만 훈련한 DLM보다 신체 부위별로 해부학적 특징이 잘 나타나는 재구성 데이터로 학습한 DLM들이 더 정확한 성능을 달성 가능하다는 것을 보여주었다. 추후 앙상블 방법을 적용한다면 더 높은 성능을 달성할 수 있을 것으로 생각된다.
Notes
References
-
Russakovsky, Olga, et al , Imagenet large scale visual recognition challenge, International journal of computer vision, (2015), 115, p211-252.
[https://doi.org/10.1007/s11263-015-0816-y]

-
Choy, Garry, et al , Current applications and future impact of machine learning in radiology, Radiology, (2018), 288(2), p318-328.
[https://doi.org/10.1148/radiol.2018171820]

-
Allen, Bibb Jr, et al. , A road map for translational research on artificial intelligence in medical imaging: from the 2018 National Institutes of Health/RSNA/ACR/The Academy Workshop, Journal of the American College of Radiology, (2019), 16(9), p1179-1189.
[https://doi.org/10.1016/j.jacr.2019.04.014]

-
Gauriau, Romane, et al , Using DICOM metadata for radiological image series categorization: a feasibility study on large clinical brain MRI datasets, Journal of digital imaging, (2020), 33, p747-762.
[https://doi.org/10.1007/s10278-019-00308-x]

-
Cho, Junghwan, et al , How much data is needed to train a medical image deep learning system to achieve necessary high accuracy?, (2015), arXiv preprint arXiv:1511.06348.
[https://doi.org/10.48550/arXiv.1511.06348]

-
Roth, Holger R., et al , Anatomy-specific classification of medical images using deep convolutional nets, 2015 IEEE 12th international symposium on biomedical imaging (ISBI), IEEE, (2015).
[https://doi.org/10.1109/ISBI.2015.7163826]

-
Bae, Kyongtae T., Intravenous contrast medium administration and scan timing at CT: considerations and approaches, Radiology, (2010), 256(1), p32-61.
[https://doi.org/10.1148/radiol.10090908]

-
Hamlin, Derek J., Burgener, F. A., Beecham, J. B., CT of intramural endometrial carcinoma: contrast enhancement is essential, American Journal of Roentgenology, (1981), 137(3), p551-554.
[https://doi.org/10.2214/ajr.137.3.551]

-
Sugimori, Hiroyuki, Classification of computed tomography images in different slice positions using deep learning, Journal of healthcare engineering, (2018), 2018.
[https://doi.org/10.1155/2018/1753480]

-
Philbrick, Kenneth A., et al , What does deep learning see? Insights from a classifier trained to predict contrast enhancement phase from CT images, American Journal of Roentgenology, (2018), 211(6), p1184-1193.
[https://doi.org/10.2214/AJR.18.20331]

-
Szegedy, Christian, et al , Inception-v4, inception-resnet and the impact of residual connections on learning, Proceedings of the AAAI conference on artificial intelligence, (2017), 31(1).
[https://doi.org/10.1609/aaai.v31i1.11231]

나 성 원
- 2017년 2월 : 건국대학교 스마트 ICT융합학과 졸업 (공학석사)
- 2022년 8월 : 건국대학교 컴퓨터공학과 졸업 (공학박사)
- 2022년 9월 ~ 현재 : 서울아산병원 의생명연구소 박사후연구원
- 주관심분야 : Deep learning, AI-based healthcare
고 유 선
- 2013년 8월 : 고려대학교 보건대학원 역학 및 보건정보학과 졸업 (석사)
- 2019년 8월 : 서울대학교 융합과학기술대학원 방사선융합의학전공 졸업 (박사)
- 2020년 1월 ~ 2022년 8월 : 서울아산병원 의생명연구소 박사후연구원
- 2022년 ~ 현재 : 울산대학교 의과대학 의학과 연구조교수
- 주관심분야 : Deep learning, AI-based healthcare, Sarcopenia, Clinical Trial, Digital Data Platform for Clinical Trial
김 경 원
- 2002년 : 서울대학교 의과대학 의학과 졸업 (학사)
- 2009년 : 서울대학교 의과대학원 의학과 졸업 (석사)
- 2012년 : 서울대학교 의과대학원 의학과 졸업 (박사)
- 2006년 ~ 2011년 : 서울대학교병원 영상의학과 전공의/임상강사
- 2012년 7월 ~ 2013년 8월 : 다나파버 암센터 하버드 의대
- 2012년 ~ 현재 : 서울아산병원 영상의학과 부교수
- 2017년 ~ 현재 : 서울아산병원 Asan Image Metrics 담당교수
- 주관심분야 : Drug development: Preclinical to clinical research, Preclinical and Clinical Application of MRI, Clinical Trial Design and Operation, Digital Data Platform for Clinical Trial