ATLAS V2.0 데이터에서 의료영상 분할 모델 성능 비교
Copyright © 2023, The Korean Institute of Broadcast and Media Engineers
This is an Open-Access article distributed under the terms of the Creative Commons BY-NC-ND (http://creativecommons.org/licenses/by-nc-nd/3.0) which permits unrestricted non-commercial use, distribution, and reproduction in any medium, provided the original work is properly cited and not altered.
초록
의료영상 공개 데이터는 수집에 한계가 있어 데이터셋의 양이 부족하다는 문제점이 있다. 때문에 기존 연구들은 공개 데이터셋에 과적합 되었을 우려가 있다. 본 논문은 실험을 통해 8개의 (Unet, X-Net, HarDNet, SegNet, PSPNet, SwinUnet, 3D-ResU-Net, UNETR) 의료영상 분할 모델의 성능을 비교함으로써 기존 모델의 성능을 재검증하고자 한다. 뇌졸중 진단 공개 데이터 셋인 Anatomical Tracings of Lesions After Stroke(ATLAS) V1.2과 ATLAS V2.0에서 모델들의 성능 비교 실험을 진행한다. 실험결과 대부분 모델은 V1.2과 V2.0에서 성능이 비슷한 결과를 보였다. 하지만 X-net과 3D-ResU-Net는 V1.2 데이터셋에서 더 높은 성능을 기록했다. 이러한 결과는 해당 모델들이 V1.2에 과적합 되었을 것으로 해석할 수 있다.
Abstract
There is a problem that the size of the dataset is insufficient due to the limitation of the collection of the medical image public data, so there is a possibility that the existing studies are overfitted to the public dataset. In this paper, we compare the performance of eight (Unet, X-Net, HarDNet, SegNet, PSPNet, SwinUnet, 3D-ResU-Net, UNETR) medical image semantic segmentation models to revalidate the superiority of existing models. Anatomical Tracings of Lesions After Stroke (ATLAS) V1.2, a public dataset for stroke diagnosis, is used to compare the performance of the models and the performance of the models in ATLAS V2.0. Experimental results show that most models have similar performance in V1.2 and V2.0, but X-net and 3D-ResU-Net have higher performance in V1.2 datasets. These results can be interpreted that the models may be overfitted to V1.2.
Keywords:
MRI Images, Neuroimaging, Stroke, Semantic SegmentationI. 서 론
최근 컴퓨터 비전의 발전으로 인해 의료영상 분야에서도 많은 비전 기술이 적용되고 있다. 그 중 영상분할(semantic segmentation)은 영상을 분석하여 질병의 위치 및 형태를 픽셀 단위로 찾아내는 기술로 다양한 연구가 진행되고 있다.
딥러닝 기반 의료 영상분할 모델 알고리즘과 모델 학습에 사용되는 데이터 셋은 최종 성능에 중요한 영향을 주는 2가지 요소이다. 특히 데이터의 품질과 양은 모델 성능에 직접적인 영향을 준다. 하지만 의료 영상 데이터는 전문가의 정밀한 어노테이션(Annotation)이 필요하며, 생명윤리위원회(Institutional Review Board)의 승인 및 임상실험자의 동의를 받아야 하기 때문에 엄청난 시간과 비용이 소모된다. 이러한 특징으로 인해 의료 영상 데이터는 수집하기 어려워 데이터 셋의 양이 작다는 문제점이 있다.
본 연구는 의료 영상 데이터 셋이 작아 기존의 모델들이 특정 데이터에 과적합(Overfitting) 되었을 가능성이 높을 것이라고 추측한다. 또한 데이터 셋의 양이 작아 성능이 낮다면 데이터 셋의 양이 증가하였을 때 성능 또한 비례하는지 변화를 알아보고자 한다. 이러한 가설들을 검증하기 위해 본 연구는 Anatomical Tracings of Lesions After Stroke(ATLAS) 데이터셋[2,15]을 사용해 성능 비교 실험을 진행하고자 한다. ATLAS 데이터셋은 영상 분할 알고리즘의 성능 비교를 위해 공개적으로 구축한 표준화된 뇌졸중 의료 영상 데이터셋이다. 과거 의료영상분할 모델의 대부분은 ATLAS를 사용해 모델의 성능 검증에 사용했다. ATLAS 데이터셋은 2018년에 ATLAS V1.2를 첫 공개했으며, 2021년 하반기에 데이터 양이 약 2배가량 증가한 ATLAS V2.0를 공개했다. 또한 공개된 시점 이후 ATLAS V2.0 데이터 셋으로 아직 검증을 실험한 연구가 없는 것이 특징이다.
때문에 본 연구에서는 ATLAS V1.2을 사용한 기존 모델로 성능을 측정하고 공개된 기존 성능과 비교함으로써 과적합이 존재하는지, 성능이 비례하게 증가하는지 가설을 검증하고자 한다. 검증을 위해 ATLAS V1.2 데이터에서 좋은 성능을 보인 8개(Unet, X-Net, HarDNet, SegNet, PSPNet, SwinUnet, 3D-ResU-Net, UNETR)의 기존 모델을 선택하여 ATLAS V2.0에 새로 추가된 데이터로 성능을 측정하는 실험을 진행했다.
실험결과 대부분의 모델들은 ATLAS V2.0에서 V1.2 보다 Dice 성능이 약 10.0%에서 29.4%만큼 증가했다. 이러한 결과는 데이터의 양이 증가할수록 모델 성능에 긍정적인 영향을 준다는 일반적인 가설에 부합하는 결과이다. 성능 지표는 성능지표 Dice와 IoU가 가장 높은 모델은 U-Net, 가장 낮은 모델은 X-net으로 나타났다. 성능이 감소한 X-net과 3D-ResU-net은 기존 데이터에 과적합 된 것이라고 추측할 수 있다.
본 연구는 다음과 같이 구성한다. 2장에서는 관련연구로 다양한 의료영상 진단 모델을 소개한다. 3장에서는 실험에서 사용하는 ATLAS 데이터 셋의 구성과 세부적인 변수 분석을 설명한다. 4장에서는 본 연구의 실험 환경과 모델의 성능을 비교하기 위한 실험 방식에 대해 서술한다. 5장에서는 연구의 실험결과를 정리하며, 6장에서는 본 연구의 결론 및 향후 연구에 대해 제시한다.
II. 관련연구
1. 기존 ATLAS에 사용된 모델 소개
Liew et al[2]은 ATLAS V1.2를 사용해 영상 분할(semantic segmentation)을 진행한 모델 DFENet, Enet, MSDF-Net, 3D U-Net, CPGAN, U-Net, D-UNet, X-net 등의 성능을 제시하였다. Ronneberger, O et al[3]은 생물의학 분야에서 이미지 분할을 목적으로 제안된 End-to-End 방식의 완전한 컨볼루션 네트워크(Fully-Convolutional Network) 기반 모델 U-Net을 개발했다. Zhou, Y et al[4]은 인코딩 단계에서 2D와 3D 컨볼루션(Convolution)을 결합한 구조를 가진 D-UNet과 Enhanced Mixing Loss(EML)라는 새로운 손실 함수를 제안했다. Qi, K et al[5]은 Feature Similarity Module(FSM)을 추가한 컨볼루션 기반 X-net을 제안했다. FSM은 장거리 의존성을 포착하기 위한 Nonlocal 연산 부분이며, 밀도가 높은 문맥상의 정보를 추출할 수 있는 장점이 있다. 본 연구에서는 코드를 공개하고 있는 U-Net, X-net을 베이스라인(baseline)으로 선정하여 실험에 사용했다.
2. 기본 segmentation 모델
영상 분할에서 우수한 성능으로 State-of-the-art(SOTA)를 달성하여 기본적으로 사용되는 Model은 HarDNet, SegNet, PSPnet 등이 있다. Badrinarayanan, V et al[6]은 길, 건물, 차량, 보행자 등 자율주행과 관련된 구조들을 Pixel-wise 영상 분할하기 위해 설계된 모델 SegNet을 제안했다. SegNet은 Encode와 Decoder로 구조로 나뉘며, Encoder Network는 VGG16의 13개 Convolution Layers를 동일하게 사용하고 있다. Zhao, H et al[7]은 기존 Fully-Convolutional Network (FCN) 모델의 단점을 개선하기 위해 PSPNet을 제안했다. PSPNet의 Pyramid Pooling Modules은 Global context information과 서로 다른 영역을 기반으로 하는 Context를 탐색할 수 있다. Chao, P et al[8]은 DenseNet에 기반한 새로운 Architecture를 구성하여 HarDNet을 제안했다. 기존 DenseNet의 Concatenation Cost를 줄이기 위해 다수의 Layer Connection들을 줄여 개선되었다.
3. Transformer 기반 모델
Dosovitskiy et al[9]는 이미지를 Patch화 하여 각 patch를 Linear Embeddings Sequence로 처리하는 Transformer 기법을 제안했다. Linear로 임베딩한 후 위치 Embedding을 더하여 이미지의 위치 정보를 활용하는 특징이 있다. Liu, Z et al[10]는 기존 Transformer 대비 계산량을 줄이면서도 다양한 규모의 이미지를 처리할 수 있는 Swin Transformer를 제안했다. 이미지 분류, 객체 인식, 영상 분할 등 여러 비전 분야에서 활발하게 사용되고 있다. Cao, H et al[11]은 Transformer 기반의 Model 모델이며 의료 분야의 영상 분할모델 Swin Unet을 제안했다. Swin Transformer 블록을 기본 단위로 사용하여 Skip연결이 있는 U자형 인코더-디코더(Encode-Decoder)구조를 가진다.
4. 3D 이미지 모델
Tomita et al[12]은 Encoder와 Decoder 구조 안에서 ResNet 블록을 결합하여 U형태로 생성한 3D-ResU-Net을 제안했다. two-stage Zoom-in&out 전략을 사용하여 작은 볼륨에서 학습한 특징을 토대로 큰 볼륨에서 모델을 미세조정 하는 특징이 있다. Hatamizadeh et al[13]는 1D Sequence-to-Sequence prediction으로 재구성한 새로운 Transformer 기반 의료영상의 영상 분할 모델 UNETR을 제안한다. UNETR은 Volumetric 데이터를 그대로 입력하는 특징이 있으며, Encoder에 Transformer를 사용하고 CNN 기반의 Decoder에 Skip Connection으로 연결하는 구조를 가진다. UNETR은 BTCV, MSD 데이터셋에서 좋은 성능을 얻었으며 BTCV 데이터셋에서 SOTA를 달성했다.
III. 데이터 셋
1. 데이터 셋 소개
ATLAS는 뇌졸중 영상 분할 (semantic segmentation)을 훈련 및 테스트하여 성능을 비교하기 위해 2018년도에 구축한 공개 데이터셋이다. 가장 먼저 공개된 데이터는 ATLAS V1.2이다. ATLAS V1.2은 299명의 환자로부터 각각 197*233 해상도의 MRI 스캔 영상을 구성한 데이터이다.
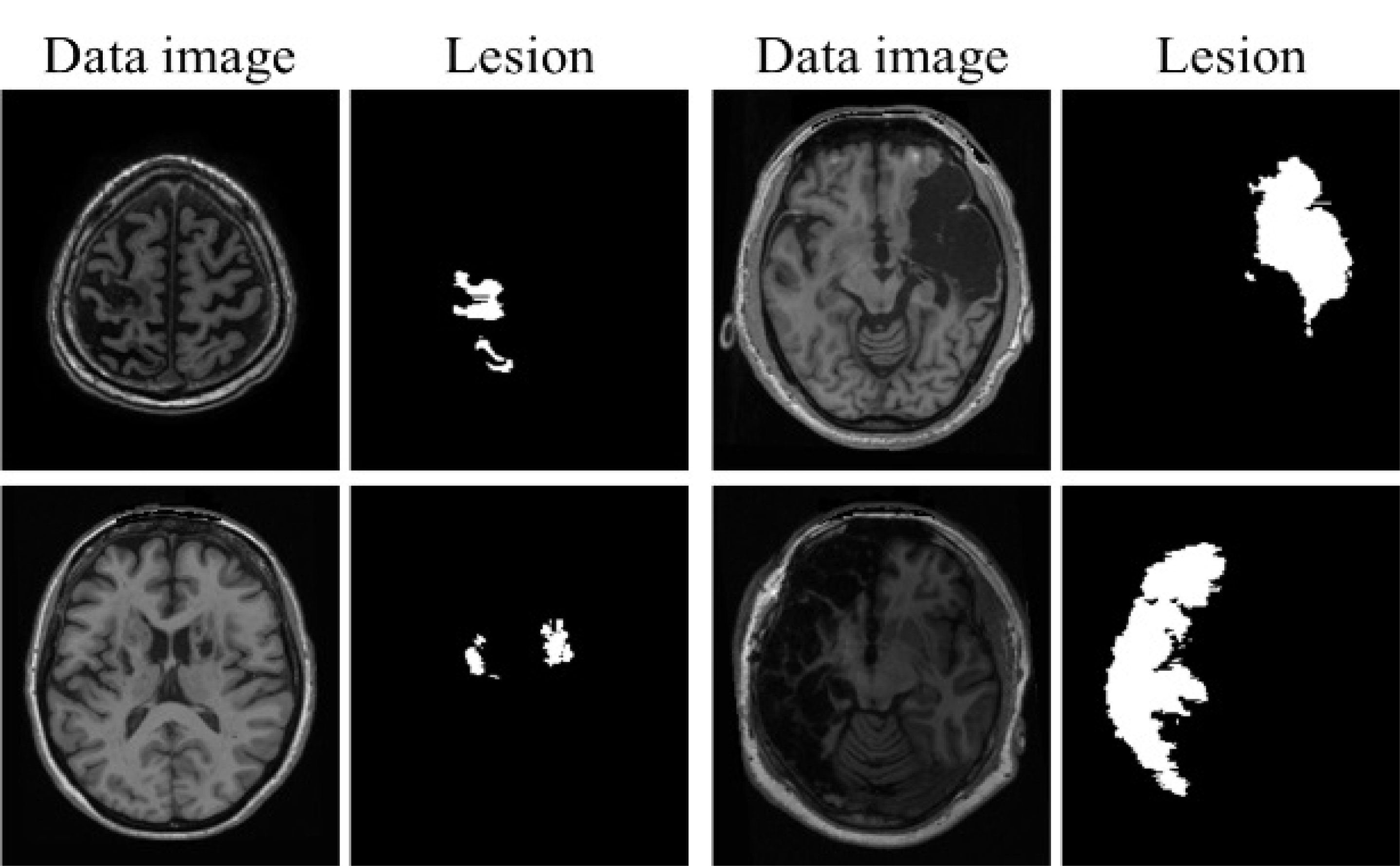
이후 2021.12에 ATLAS V2.0 이 발표되었다. 데이터의 혼동을 줄이고자 V1.2와 V2.0으로 ATLAS의 Type을 구분하여 명명한다. V2.0은 655명의 환자정보로 구성된 훈련 데이터 셋과 300명의 환자정보로 구성된 테스트 데이터 셋으로 이루어져 있다. 현재 훈련 데이터 셋은 Ground Truth(GT)가 공개되었으나, 테스트 데이터 셋은 공개 경진대회 진행으로 인해 GT가 미공개 상태이다. 그림 1은 V2.0의 이미지와 Mask의 예시이다. Mask로 표기된 부분이 병변(Lesion)을 의미한다.
2. 데이터 셋 구성
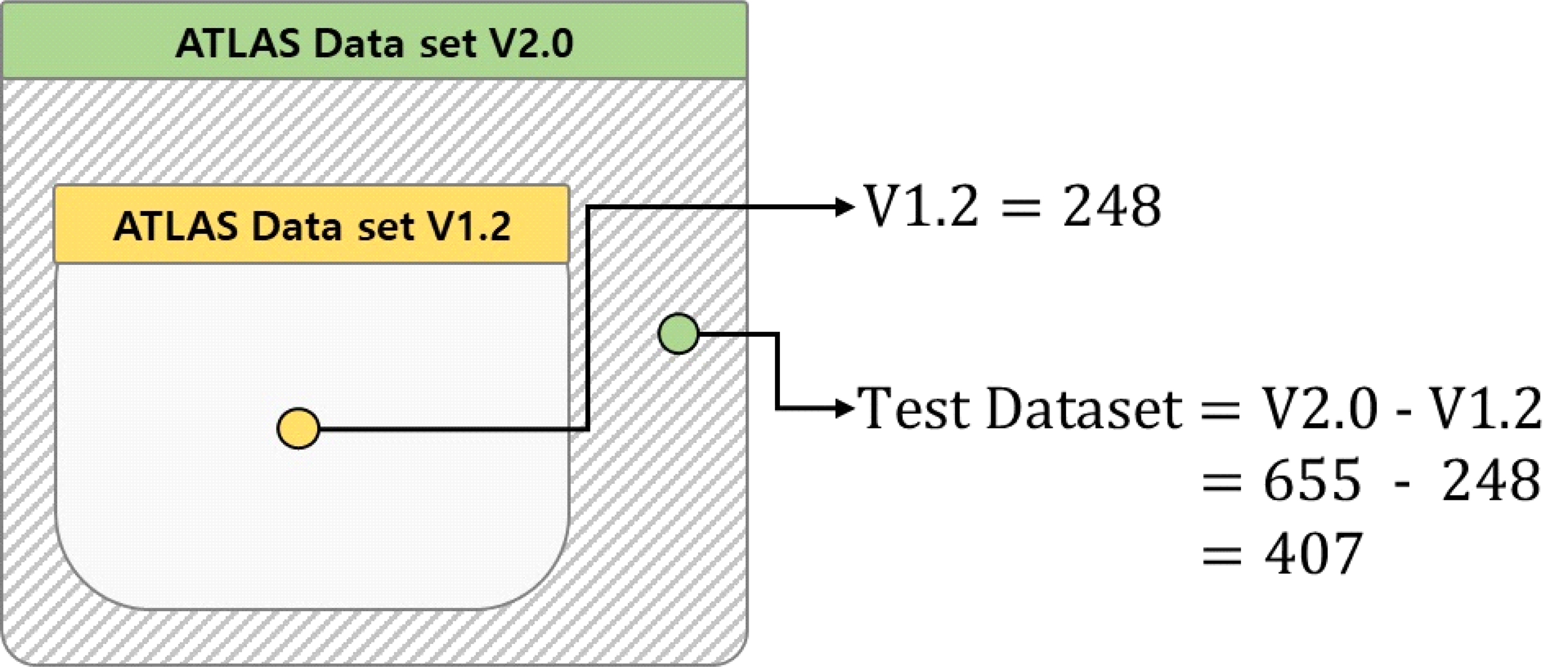
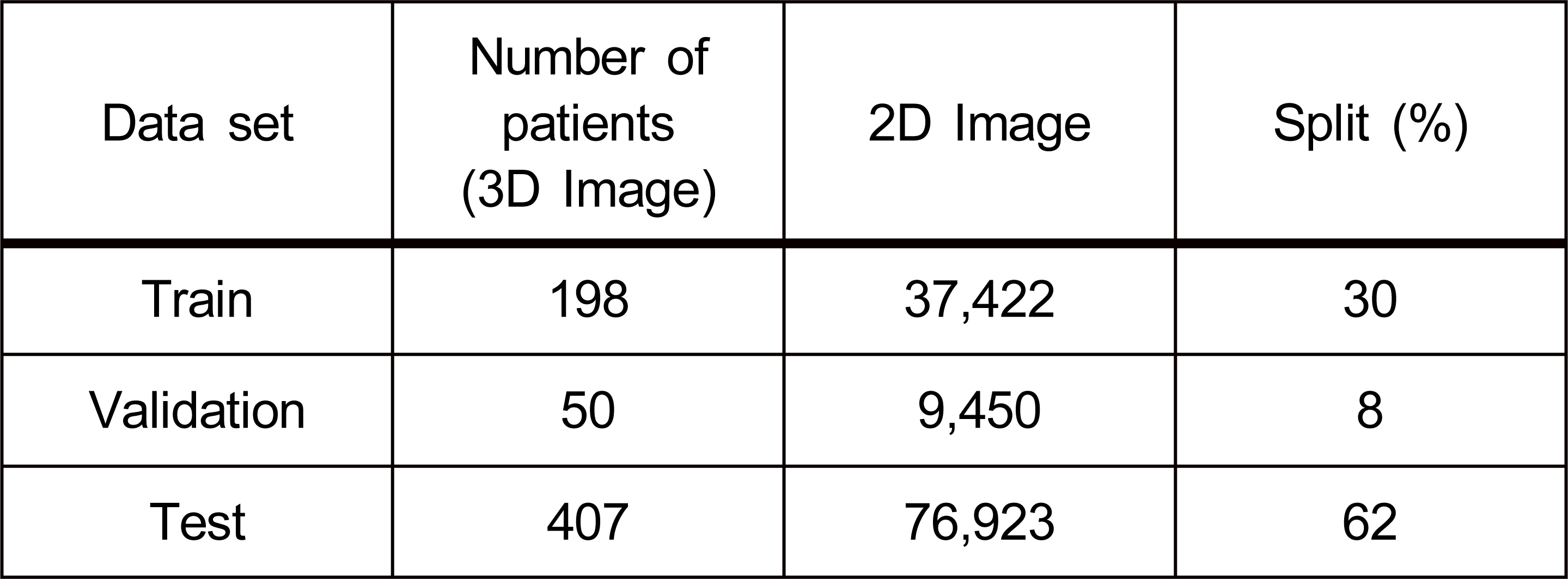
본 연구에서는 V2.0 데이터가 재 생성된 데이터가 아닌 실제 환자의 정보로 만들어진 데이터이기 때문에 훈련 데이터셋의 양으로도 과적합이 발생하지 않을 수 있는 연구를 진행할 수 있다고 판단했다. 따라서 655명의 환자정보로 구성된 훈련 데이터셋을 실험의 전체 데이터셋으로 구성하여 실험을 진행한다. 그림 2와 같이 실험에 사용하는 V2.0 데이터 셋 안에는 기존의 V1.2 데이터가 포함되어 있다. 베이스라인과 같은 버전의 데이터로 모델을 학습하고, V2.0에서 새롭게 추가된 데이터를 테스트 데이터 셋으로 사용하기 위해 본 연구는 V2.0에서 V1.2 데이터를 분리하여 훈련과 테스트 데이터 셋으로 분류한다. 최종데이터 셋은 248명의 환자 정보가 포함된 훈련 데이터셋과 407명의 환자 정보가 포함된 테스트 데이터셋으로 이루어져 있다.
3. 데이터 구성 분석
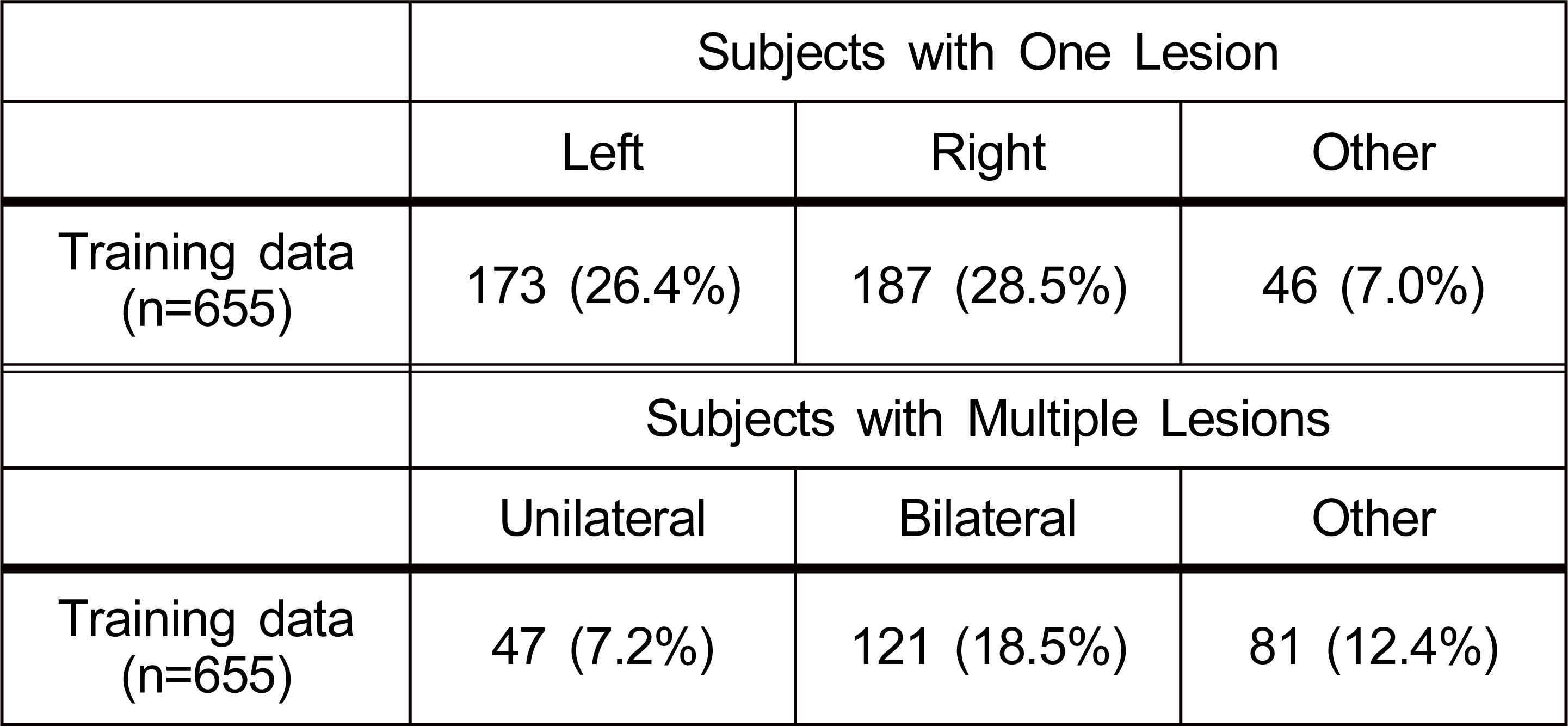
ATLAS 실험을 진행하는 데이터 셋의 병변의 분포를 알아보기 위해 단일 병변과 다발성 병변의 빈도와 위치를 분석한다. V2.0에서는 환자의 59.9%가 단일 병변을, 38.1%는 다발성 병변을 가진다. 병변은 다른 병변과 인접하지 않은 경우 별도로 추가하여 계산했다. 표 1은 Liew et al[2]이 병변 빈도에 따라서 우반구 및 좌반구로 나누어 환자를 집계한 표이다. 병변은 좌반구와 우반구 사이에 거의 균등하게 분포되어 있었으며, 피험자의 57.1 %는 적어도 하나의 좌반구 병변을, 58.8 %는 하나의 우반구 병변을, 22.9 %는 소뇌 또는 뇌간에 하나의 병변을 보였다.
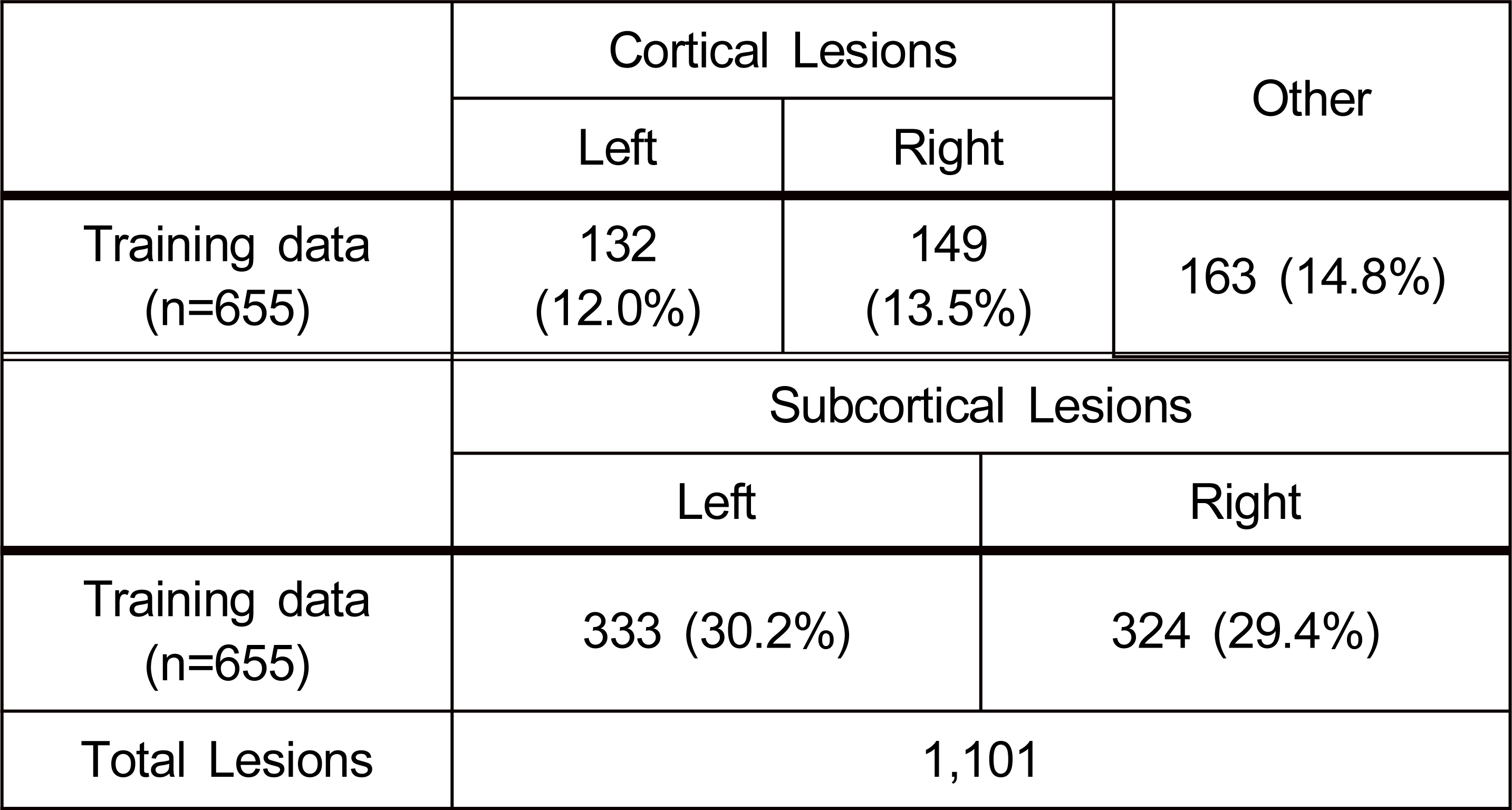
표 2는 Liew et al[2]가 병변이 존재하는 위치에 따라서 환자 수를 분류한 표이다. 병변의 위치는 Cortical Lesions에 25.5%, Subcortical Lesions에 43.0% 존재했다. Subcortical Lesions에 가장 많은 병변이 분포되어 있으며, 그 다음 Cortical Lesions와 기타 순서로 나타난다. 또한 병변은 Subcortical Lesions과 Cortical Lesions의 좌우에 균등하게 분포한다.
IV. 실험 설계
1. 실험 평가지표 및 환경
본 연구에서는 실험의 성능 평가 지표로 Dice와 Intersection Over Union (IoU)를 사용한다. Dice는 의료 영상 분할의 성능을 측정하는 정량적 평가 방법 중 하나로, 예측한 영역과 실제 GT가 얼마나 일치하는지 그 비율을 나타내는 지표이다. 예측한 영역과 GT의 겹쳐지는 영역을 2로 곱한 후, 두 이미지의 총 픽셀 수로 나누어 정의할 수 있다. 계산 공식은 (1)과 같다. IoU는 영상 분할 분야에서 가장 흔하게 쓰이는 메트릭이다. 예측된 분할 map과 GT 사이에 교차 영역을 예측된 분할 map과 GT 사이의 결합 영역(union)으로 나누어 산정한다. 계산 공식은 (2)와 같다.
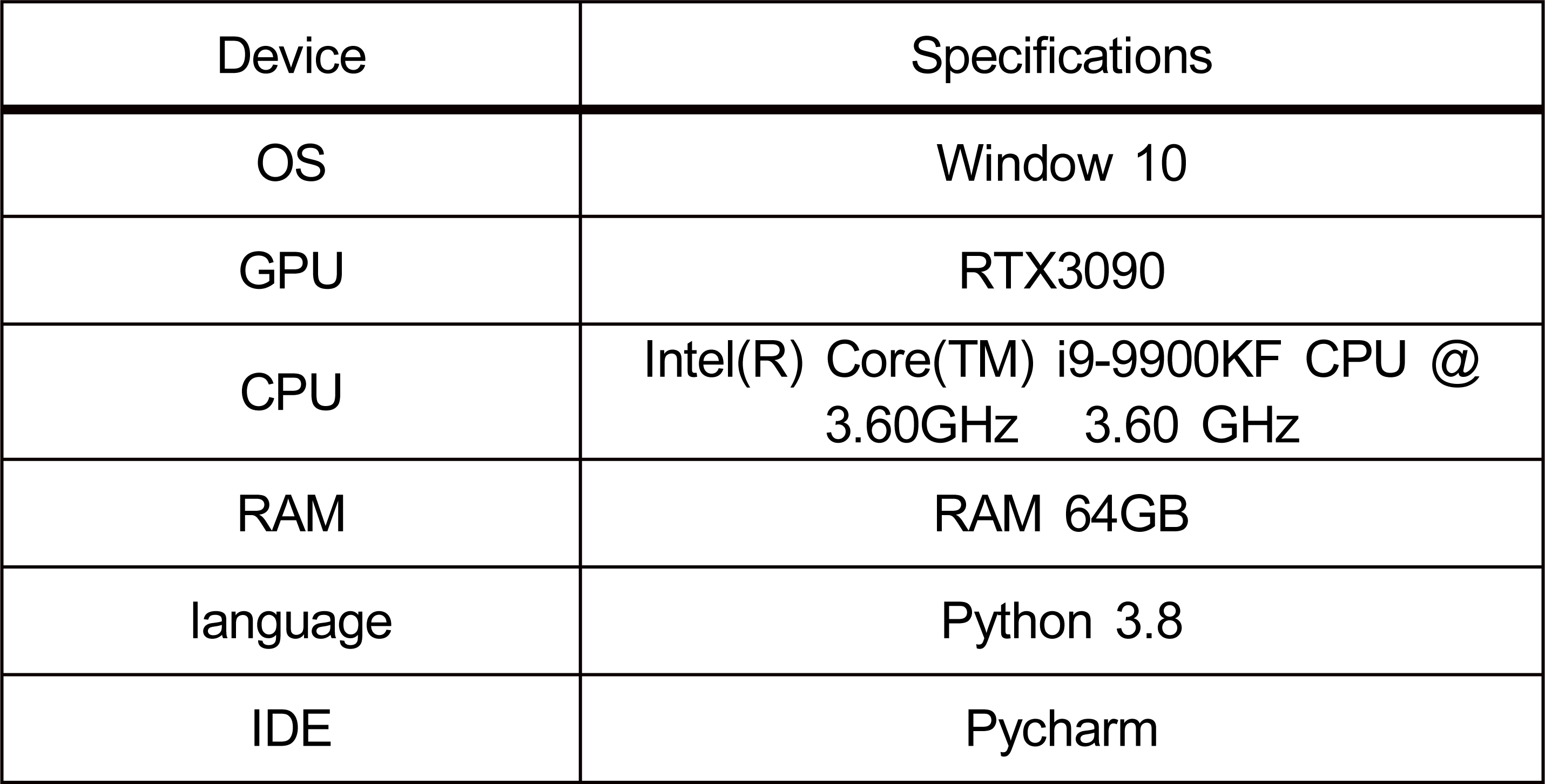
공식(1), (2)에서 TP, TN, FP, FN은 Confusion matrix의 True Positive(TP), True Negative(TN), False Positive(FP), False Negative(FN)를 의미한다. 표 3은 본 연구에서 사용한 실험 환경이다.
2. 실험 설계
본 연구의 실험 목적은 V1.2를 학습하여 실험을 진행한 모델들이 데이터 양이 증가한 V2.0에서 성능 지표가 어떻게 변화하는지, 기존 데이터에 과적합 되었는지 알아보기 위함이다. 실험 데이터로 사용하는 ATLAS는 인위적으로 생성된 데이터가 아닌 실제 환자의 MRI 영상에서 뇌출혈 위치를 픽셀 단위로 표기한 데이터이다. 때문에 기존 모델들이 실제 환자의 뇌졸중 진단에 여전히 비슷한 성능을 유지하는지 평가할 수 있을 것이라고 판단한다. 다양한 모델들의 성능평가를 위해 본 연구에서는 CNN기반과 Transformer기반 등 8개의 영상 분할 모델을 베이스라인으로 선정하여 실험을 진행했다.
실험에 사용하는 전체 데이터 셋은 655명의 환자 정보로 구성된다. 1명의 3D 환자 정보는 189장의 2D 이미지 Slice가 나열되어 있는 형태이다. 따라서 전체 데이터 셋을 2D 이미지로 변경한 결과는 총 123,795장이다. 표 4는 실험에 사용한 데이터 셋을 나눈 결과이다. 훈련 데이터 셋은 V1.2의 198명 환자 데이터 37,422장 이미지를 사용한다. 검증 데이터 셋은 V1.2의 50명 환자 데이터 9,450장 이미지를 사용한다. 테스트 데이터 셋은 V2.0에 새롭게 포함된 407명의 환자 데이터 76,923장 이미지를 사용한다.
3. 모델 학습
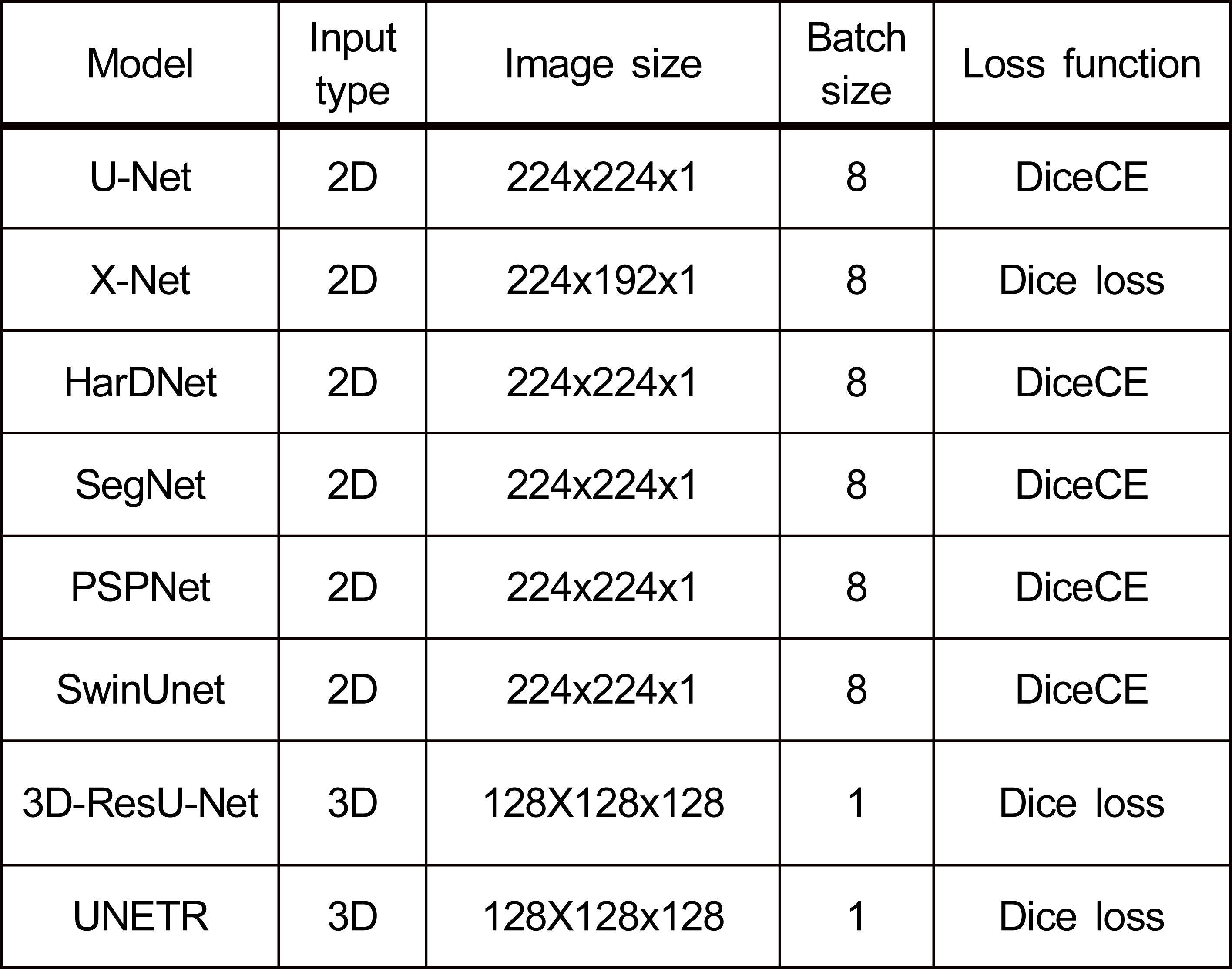
본 연구에서는 2장 관련연구에서 소개한 의료영상분할 모델 중 코드를 제공하고 있거나, 우수한 성능을 달성한 모델 8개를 선택해 다시 학습하고 성능 비교 실험을 진행했다. 표 5는 모델 학습에 사용한 파라미터 설정을 정리한 결과이다. 모델의 입력 데이터는 입력 이미지 형태에 따라 2D와 3D로 나뉜다. Optimizer는 모두 Adam을 적용했다. 학습 진행 중 과적합을 방지하기 위해 10번의 에포크 안에서 검증 손실 값이 이전 검증 손실 값 보다 감소하지 않을 시 Early stopping으로 훈련의 조기 종료를 진행했다. 손실 함수는 Dice 손실함수와 DiceCE 두 개를 사용한다. Dice 손실함수는 Dice score를 1로 뺀 값이다. DiceCE는 교차 엔트로피 함수(Cross entropy loss)와 Dice의 손실함수를 합산하여 사용하는 손실 함수이다.
V. 실험 결과
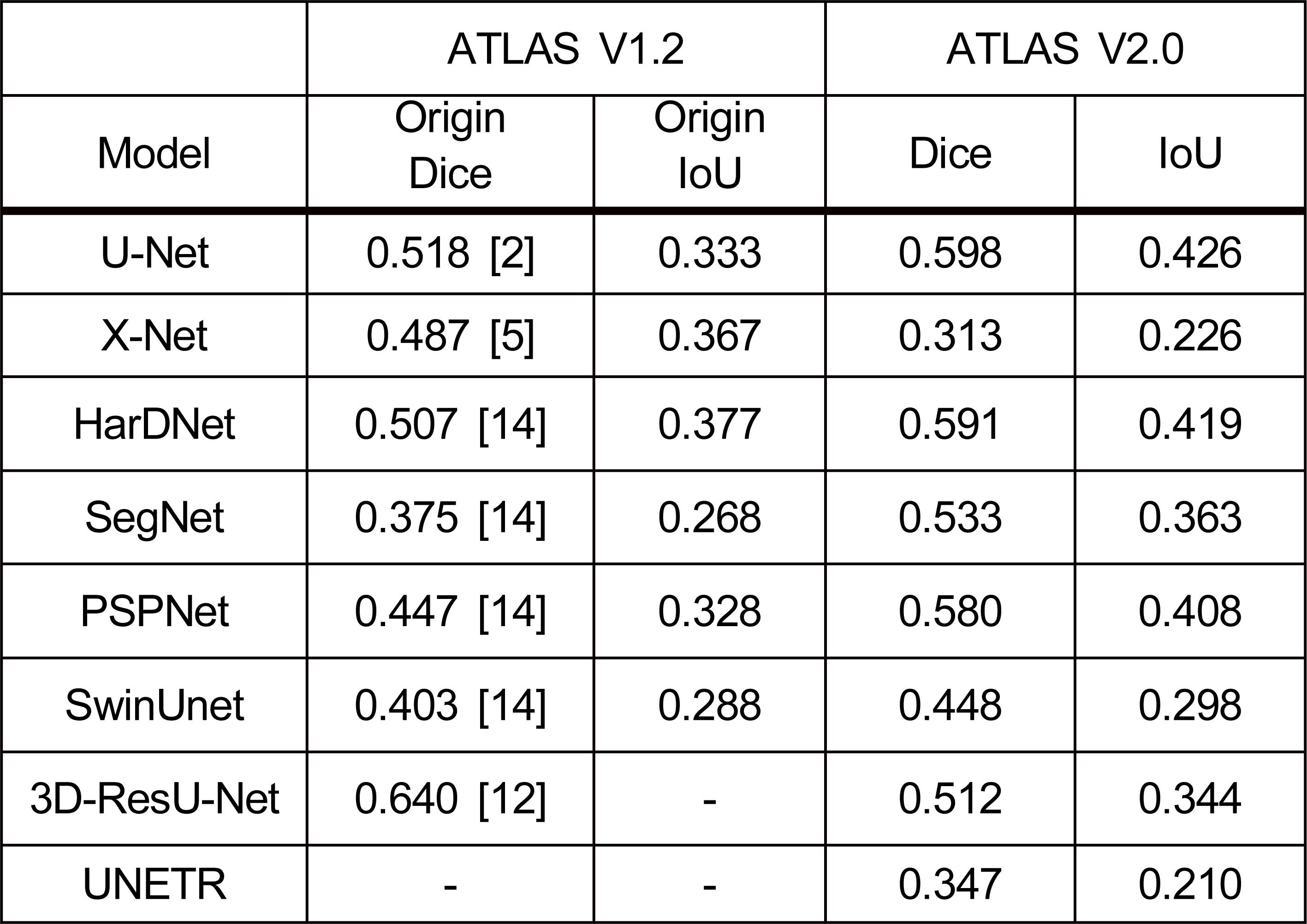
표 6은 성능 비교 실험을 진행한 결과이다. Origin Dice와 Origin IoU V1.2 데이터로 학습과 테스트를 진행한 Dice와 IoU 성능 결과이다. Origin Dice와 Origin IoU는 기존 모델들의 문헌에서 참고하여 표 6에 기재했다.
U-Net, HarDNet, SegNet, PSPNet, SwinUnet은 V2.0를 사용한 실험에서 더 높은 Dice와 IoU 성능을 보였다. V2.0를 사용한 실험에서 성능이 증가한 모델들은 V1.2 대비 최소 10.0%에서 최대 29.6% 성능이 증가했다. V1.2에서 Dice와 IoU 성능이 가장 높은 모델은 3D-ResU-Net이지만, 본 연구에서 성능이 가장 높은 모델은 U-Net이다. 기존 성능 보다 Dice와 IoU가 가장 많이 상승한 모델은 SegNet으로 Dice가 0.158, IoU가 0.095 상승했다. X-net은 기존 성능보다 Dice가 -0.174로 가장 크게 하락했으며, 실험에서 가장 낮은 Dice를 얻었다. UNETR은 기존 성능이 없으나 동일한 3D 모델인 3D-ResU-Net에 비해 성능이 낮게 나왔다.
VI. 결론
본 연구는 의료영상 데이터의 양이 작은 고질적인 문제점 때문에 V1.2기반 모델이 데이터에 과적합 되었을 가능성이 있다고 가정했다. 가설검증과 함께 데이터가 증가했을 경우 기존 모델들의 성능이 어떻게 변화할지 알아보고자 실험을 진행했다.
실험 결과 X-net과 3D-ResU-net을 제외한 6가지 영상분할 모델의 성능이 증가한 것을 알 수 있다. 이것은 데이터가 증가할수록 영상분할 모델 성능에 긍정적인 영향을 준다는 일반적인 가설에 부합하는 결과이다. 하지만 성능이 감소한 영상분할 모델 또한 존재했다. 성능이 감소한 X-net과 3D-ResU-net은 기존 데이터에 과적합 된 것이라고 추측할 수 있다. 또한 영상분할 모델들의 성능 증가 수치의 차이를 통해 성능이 더 높게 증가했을수록 더욱 일반화된 영상분할 모델이라고 평가할 수 있었다.
데이터의 양이나 속성에 변화가 생길 경우 모델 성능 또한 함께 변화한다는 결과를 통해 본 연구는 데이터의 변동성에도 능동적인 영상분할 모델을 만들기 위한 새로운 접근법의 필요성을 제시한다.
Notes
References
-
Zhang, S., Xu, S., Tan, L., Wang, H., Meng, J., Stroke Lesion Detection and Analysis in MRI Images Based on Deep Learning, Journal of Healthcare Engineering, (2021, Apr.), 2021.
[https://doi.org/10.1155/2021/5524769]

-
Liew, S.-L., et al. , A large, curated, open-source stroke neuroimaging dataset to improve lesion segmentation algorithms, Scientific Data, Springer Science and Business Media LLC, (2022, Jun.), 9(1).
[https://doi.org/10.1038/s41597-022-01401-7]

-
Ronneberger, O., Fischer, P., Brox, T., U-Net: Convolutional Networks for Biomedical Image Segmentation, Lecture Notes in Computer Science, Springer International Publishing, (2015, October), p234-241.
[https://doi.org/10.1007/978-3-319-24574-4_28]

-
Zhou, Y., Huang, W., Dong, P., Xia, Y., Wang, S., D-UNet: A Dimension-Fusion U Shape Network for Chronic Stroke Lesion Segmentation, IEEE/ACM Transactions on Computational Biology and Bioinformatics, Institute of Electrical and Electronics Engineers (IEEE), (2021, May), 18(3), p940-950.
[https://doi.org/10.1109/TCBB.2019.2939522]

-
Qi, K., et al. , X-Net: Brain Stroke Lesion Segmentation Based on Depthwise Separable Convolution and Long-Range Dependencies, Lecture Notes in Computer Science, Springer International Publishing, (2019, October), p247-255.
[https://doi.org/10.1007/978-3-030-32248-9_28]

-
Badrinarayanan, V., Kendall, A., Cipolla, R., SegNet: A Deep Convolutional Encoder-Decoder Architecture for Image Segmentation, IEEE Transactions on Pattern Analysis and Machine Intelligence, Institute of Electrical and Electronics Engineers (IEEE), (2017, Dec.), 39(12), p2481-2495.
[https://doi.org/10.1109/TPAMI.2016.2644615]

-
Zhao, H., Shi, J., Qi, X., Wang, X., Jia, J., Pyramid Scene Parsing Network, 2017 IEEE Conference on Computer Vision and Pattern Recognition (CVPR), IEEE, (2017, Jul.).
[https://doi.org/10.1109/cvpr.2017.660]

-
Chao, P., Kao, C.-Y., Ruan, Y., Huang, C.-H., Lin, Y.-L., HarDNet: A Low Memory Traffic Network, 2019 IEEE/CVF International Conference on Computer Vision (ICCV), IEEE, (2019, Oct.).
[https://doi.org/10.1109/iccv.2019.00365]

-
Dosovitskiy, A., et al. , An Image is Worth 16x16 Words: Transformers for Image Recognition at Scale, (2020), arXiv.
[https://doi.org/10.48550/arXiv.2010.11929]

-
Liu, Z., et al. , Swin Transformer: Hierarchical Vision Transformer using Shifted Windows, Proceedings of the IEEE/CVF International Conference on Computer Vision (ICCV), (2021), p10012-10022.
[https://doi.org/10.48550/arXiv.2103.14030]

-
Cao, H., et al. , Swin-Unet: Unet-like Pure Transformer for Medical Image Segmentation, (2021), arXiv.
[https://doi.org/10.48550/arXiv.2105.05537]

-
Tomita, N., Jiang, S., Maeder, M. E., Hassanpour, S., Automatic post-stroke lesion segmentation on MR images using 3D residual convolutional neural network, NeuroImage: Clinical, Elsevier BV, (2020), 27, p102276.
[https://doi.org/10.1016/j.nicl.2020.102276]

-
Hatamizadeh, Ali, et al. , Unetr: Transformers for 3d medical image segmentation, Proceedings of the IEEE/CVF Winter Conference on Applications of Computer Vision, (2022).
[https://doi.org/10.48550/arXiv.2103.10504]

-
Gu, Y., Piao, Z., Yoo, S. J., STHarDNet: Swin Transformer with HarDNet for MRI Segmentation, Applied Sciences, MDPI AG, (2022, Jan.), 12(1), p468.
[https://doi.org/10.3390/app12010468]

- Anatomical Tracings of Lesions After Stroke (ATLAS) R1.1, Available online: http://fcon_1000.projects.nitrc.org/indi/retro/atlas.html accessed on 15 Feb 2021.
우 소 연
- 2021년 ~ 현재 : 세종대학교 인공지능학과 석사과정
- 2020년 : 을지대학교 식품산업외식학과 졸업(학사)
- 주관심분야 : 데이터 마이닝, 기계학습, 인공지능, 메타러닝
구 영 현
- 2022년 ~ 현재 : 세종대학교 인공지능학과 교수
- 2014년 ~ 2022년 : 세종대학교 전임연구교수
- 2006년 ~ 2014년 : 세종대학교 컴퓨터공학과 졸업(박사)
- 2006년 : 세종대학교 컴퓨터공학과 졸업(석사)
- 2004년 : 세종대학교 컴퓨터소프트웨어학과 졸업(학사)
- 주관심분야 : 데이터 마이닝, 기계학습, 인공지능, 메타러닝
유 성 준
- 2002년 ~ 현재 : 세종대학교 컴퓨터공학과 교수
- 2000년 ~ 2002년 : (주)서치캐스트 연구소장
- 1998년 ~ 2000년 : 한국전자통신연구원 정보검색연구팀 팀장
- 1996년 : Syracuse University 전산학과졸업(박사)
- 1990년 : 고려대학교 전자공학과 졸업(석사)
- 1982년 : 고려대학교 전자공학과 졸업(학사)
- 주관심분야 : 러닝 시스템, 패턴 인식, 데이터마이닝, 인공지능