딥러닝 기반 의료 영상 병변 세분화를 위한 픽셀 단위 손실 함수 성능 비교
Copyright © 2025 Korean Institute of Broadcast and Media Engineers. All rights reserved.
“This is an Open-Access article distributed under the terms of the Creative Commons BY-NC-ND (http://creativecommons.org/licenses/by-nc-nd/3.0) which permits unrestricted non-commercial use, distribution, and reproduction in any medium, provided the original work is properly cited and not altered.”
초록
딥러닝 모델 기반 정확한 병변 영역 분할을 위해서는 영상 유형과 모델 특성에 부합하는 손실 함수를 선택해야 한다. 본 연구에서는 10개 픽셀 단위 손실 함수를 피부, 내시경, 초음파를 포함한 6개 의료 영상 데이터셋과 12개 딥러닝 모델에 대해 반복 실험하여 종합적으로 비교 분석하였다. 모든 데이터셋과 모든 모델에서 최고 정확도를 보이는 손실 함수는 없었으나, 복합 손실 함수가 단일 손실 함수보다 정확도가 전반적으로 우수했다. 특히, IoU와 Focal 손실을 결합한 IoUFocal 손실 함수는 Dice 정확도 순위에 따라 성능을 점수화 하였을 때, 데이터셋과 모델 모두에서 가장 높은 종합 점수를 보였다. 시각화 분석 결과, 정확도가 높은 손실 함수일수록 병변이 아닌 영역에서의 예측 노이즈가 감소하는 경향을 확인하였다. 본 연구의 종합적 비교 분석은 연구자들이 각자의 데이터셋과 모델에 최적화된 손실 함수를 선택하는 데 있어 유의미한 지침을 제공할 것으로 기대된다.
Abstract
Accurate lesion segmentation based on deep learning requires selecting loss functions that align with both the image type and model characteristics. In this study, we conducted repeated experiments with 12 deep learning models on six medical imaging datasets (skin, endoscopy, ultrasound), comparing 10 pixel-based loss functions. Although no single loss function was universally accurate across all datasets and models, compound loss functions demonstrated generally superior accuracy compared to individual loss functions. Notably, when performance was evaluated based on Dice accuracy ranking, the IoUFocal loss function (a combination of IoU and Focal losses) achieved the highest overall score across both datasets and models. Furthermore, visualization analysis indicated that loss functions with higher accuracy tended to reduce prediction noise in non-lesion areas. This comprehensive comparative study is expected to provide valuable guidelines for researchers in selecting loss functions optimized for their specific datasets and models.
Keywords:
Lesion Medical Images, Image Segmentation, Loss Function, Deep Learning, Accuracy ComparisonⅠ. 서 론
병변 영역 분할(lesion segmentation)은 의료 영상에서 상처, 염증, 용종 등 병변 영역을 검출하는 이미지 분할(image segmentation)의 한 연구 주제이다[1]. 병변 영역 분할은 의료 영상 데이터를 통한 질병의 진단, 치료 전략 수립, 예후 평가에 중요하게 활용된다[2]. 최근 딥러닝 기반 인공지능 모델은 의료 영상 분할 분야에서 뛰어난 성능을 보여주고 있다[3]. 딥러닝 모델은 병변 영역 분할의 학습 과정에서 병변 이미지와 예측한 이미지의 픽셀 일치 여부를 수식을 통해 수치화하여 사용하며, 이 수식을 손실 함수(loss function)라고 한다. 딥러닝 모델은 이 손실 함수 값이 최소화되도록 학습을 수행하기 때문에, 예측 정확도는 손실 함수에 직접적으로 영향을 받는다. 따라서, 손실 함수의 특성을 이해하고 데이터셋과 모델의 특성에 맞추어 적절하게 손실 함수를 선택하는 것은 딥러닝 모델의 예측 정확도 성능을 향상에 있어 중요한 한 주제이다[4].
이미지 분할 딥러닝에서 손실 함수는 일반적으로 병변 정답 라벨링 이미지와 병변 예측 이미지를 픽셀 일치 여부를 비교하여 계산하는 픽셀-to-픽셀로 손실 함수가 사용된다. 이러한 손실 함수는 지역 기반 손실(region-based loss)과 분포 기반 손실(distribution-based loss)로 구분한다[4]. 지역 기반 손실은 예측된 영역 픽셀과 실제 영역 픽셀의 겹치는 정도를 측정하며, IoU(Intersection over Union) 손실과 Dice 손실이 있다. 분포 기반 손실은 예측된 영역 픽셀의 출력 가중치의 크기와 실제 영역 픽셀의 사이 차이를 계산하며, 크로스 엔트로피(Cross entropy) 손실과 포컬(Focal) 손실이 있다. 또한, 이러한 단일 손실 함수들을 조합하여 만들어낸 새로운 손실 함수를 복합 손실(compound loss)이라고 한다. 다양한 일반 이미지 데이터셋에서 손실 함수들이 최고 성능을 보여주는 경향을 보여주었다[4,5].
최근 연구는 의료 이미지 데이터셋에 대해서 손실 함수의 성능을 비교하였다[5]. 해당 연구에서는 CT 이미지 폐암 병변, CT 이미지 췌장암 병변 데이터셋의 두 종류 데이터셋과 U-Net 딥러닝 모델이라는 한 종류 알고리즘에 대해서 비교 실험을 수행하여 복합 손실 함수가 단일 손실 함수보다 우수한 성능을 보인다고 보고하였다. 그런데, 해당 연구는 초음파 유방암, 피부 궤양, 내시경 위암 등 다양한 종류의 의료 데이터셋과, 다양한 딥러닝 모델에 대한 종합적인 성능 비교를 수행하여 결론을 제시하지는 못하였다는 한계가 있다[5]. 본 연구에서는 6종류의 병변 이미지 데이터셋과 12종류의 딥러닝 모델에 대해서 10종류의 픽셀 단위 손실 함수를 반복실험하여 병변 영역 분할에 대한 종합적인 비교 분석을 수행하는 것을 목표로 한다.
Ⅱ. 실험 설계
1. 데이터셋
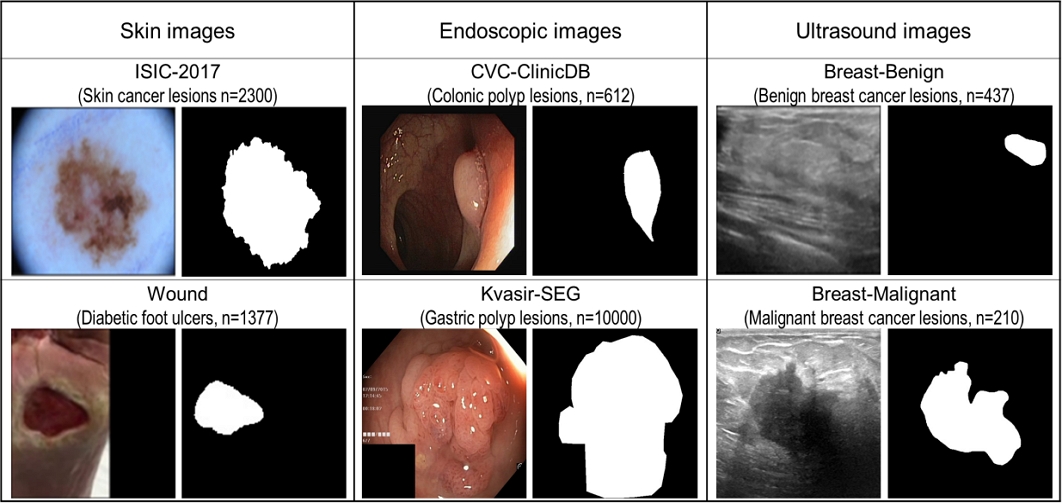
본 연구에서는 6종의 병변 영역 분할 공개 이미지 데이터셋을 사용하였다. 이 데이터셋들은 영상의 종류별로 피부암 병변(ISIC-2017), 족부 궤양 병변(Wound)은 피부 사진에 대한, 대장 용종 병변(CVC-ClinicDB)과 위장 용종 병변(Kvasir-SEG)은 내시경 영상에 대한, 유방암 양성 용종 병변(Breast-Benign)과 유방암 음성 용종 병변(Breast-Malignant)은 초음파 영상에 대한 데이터셋으로 구분할 수 있다. 각 데이터셋은 원본 이미지와 병변 영역 라벨 이미지의 쌍들로 구성된다. 그림 1은 6종류의 이미지 데이터셋의 예시와 이미지 개수를 보여준다.
2. 딥러닝 모델
실험을 위한 딥러닝 모델로는 의료 영상 분할 연구 분야에 초기부터 최신까지의 대표적인 12개의 딥러닝 모델을 선정하여 비교하였다. 각 모델은 일반 이미지 모델, 의료 이미지 모델, 특정 의료 이미지 특화 모델로 구분할 수 있다.
- 일반 이미지 모델 :
- ① FCN (Fully Convolutional Network)[6]
- ② DeepLab-V3+[7]
- 의료 이미지 모델 :
- ③ U-Net[8], ④ U-Net++[9]
- 특정 의료 이미지 특화 모델 :
- ⑤ ColonSegNet[10], ⑥ FAT-Net[11], ⑦ FCBFormer[12]
- ⑧ ColonFormer[13], ⑨ CaraNet[14], ⑩ ESFPNet[15]
- ⑪ DuckNet[16], ⑫ EMCADNet[17]
각 모델의 구조는 원 저자의 논문을 따라 구현하여 수행되었다. 본 연구의 주제는 손실 함수이기 때문에 모델 구조에 설명은 생략하고, 참고 논문으로 표기하였다.
3. 손실 함수
본 연구에서는 비교한 10종류의 픽셀-to-픽셀 손실 함수는 지역 기반 손실, 분포 기반 손실, 복합 손실로 나뉠 수 있다.
지역 기반 손실은 예측 영역의 병변 픽셀의 집합(A={Ai,Ai는 예측 이미지에서 병변으로 예측된 픽셀})과 라벨링 영역의 픽셀(B={Bi, Bi는 라벨 이미지에서 병변으로 라벨링된 픽셀})의 일치도를 측정하며, 그 종류는 IoU 손실과 Dice 손실이 있다.
- ① IoU 손실 (IoU Loss)
- ② Dice 손실 (Dice Loss)
분포 기반 손실은 예측 이미지의 각 픽셀에 대한 가중치 출력(pi := 예측 이미지에서 i번째 픽셀이 병변인지에 대한 가중치 출력 값)과 라벨링 이미지의 각 픽셀의 라벨(Bi := 라벨 이미지에서 i번째 픽셀이 병변인지 라벨 값, 병변이면 1, 아니면 0)의 유사성을 측정하며, 그 종류는 이진 크로스 엔트로피 손실과 포컬 손실이 있다.
- ③ 이진 크로스 엔트로피 손실 (Binary Cross Entropy Loss)
- ④ 포컬 손실 (Focal Loss)
포컬 손실에서 α는 양성 클래스를 강조하기 위한 값이고, γ는 값이 커질수록 어려운 예제에 기여도를 상대적으로 높여줌으로써 어려운 예제에 집중하도록 조정하는 값이다. 본 실험에서는 기존 연구[3]를 따라 α=0.8, γ=0.2를 사용하였다.
복합 손실 함수는 지역 기반 및 분포 기반 손실을 결합한 것이다. 예를 들어, IoUDice Loss는 IoU Loss와 Dice Loss를 합한 값이다. 우리는 다음과 같은 이진 조합의 복합 손실 함수를 비교하였다: ⑤ IoUDice Loss ⑥ IoUBCE Loss ⑦ IoUFocal Loss ⑧ DiceBCE Loss ⑨ DiceFocal Loss ⑩ BCEFocal Loss를 비교하였다.
4. 실험 설계 및 환경
딥러닝 모델의 입력 이미지 크기는 224×224픽셀로 크기 조절하여 동일하게 전처리하였다. 데이터 증강은 수행하지 않았다. 각 손실 함수에 대해서 6종류의 데이터셋, 10종류의 딥러닝 모델별로 각 5회씩 반복 실험을 수행하여 손실 함수의 성능을 평가하였다. 각 반복 실험에서 데이터셋은 학습:검증:시험=60:20:20 비율로 랜덤 분할하여 실험을 진행하였다. 각 실험에서는 배치 크기 8, 옵티마이저 Adam, 학습률 0.001, 최대 에폭 200, 고정된 랜덤 시드 42, 과적합 방지를 위해서 검증 데이터셋의 Dice 지표에 대해 patience 40의 조기 종료(Early Stopping)를 적용하여 딥러닝 모델을 학습하였다. 각 실험에서는 시험 데이터셋에 대해서 Dice, IoU, 재현율(Recall), 정밀도(Precision)의 4개 정확도 성능 지표를 계산하고 평균한 값으로 평가하였다. 각 실험은 i9-13900KF CPU, 64G DDR5 RAM, RTX-4090 GPU 하드웨어 환경과 python 3.12.7, torch 2.4.1, cuda 12.1 소프트웨어 버전에서 수행되었다.
Ⅲ. 실험 및 결과
1. 종합 성능 비교
6종의 병변 이미지 데이터셋과 12개의 딥러닝 모델을 활용하여 10종류의 손실 함수에 대해 5회 반복 실험하고 Precision, Recall, IoU, Dice 정확도의 평균을 구하여 시각화하였다(그림 2A). 모든 평가 지표에서 우세한 특정한 성능 지표는 관찰되지 않았다. 다만, Focal 손실은 4종류의 정확도에서 모두 차이가 나타날 정도로 최하 등수를 기록함으로써 명확하게 정확도 성능이 낮은 모습을 보여주었다. DiceBCE나 IoUFocal가 평균 Dice 정확도에서 1, 2등을 각각 차지함으로써, 기존 연구의 두 가지 손실 함수를 조합한 복합 손실이 개별 손실보다 우세하다는 결과를 보여주었다[5]. 복합 손실 중에서 IoUFocal은 Dice, IoU, Precision, Recall 정확도에서 각각 2, 2, 3, 2등을 각각 기록하여서, 종합적으로 상위 정확도 성적을 보여주었다.
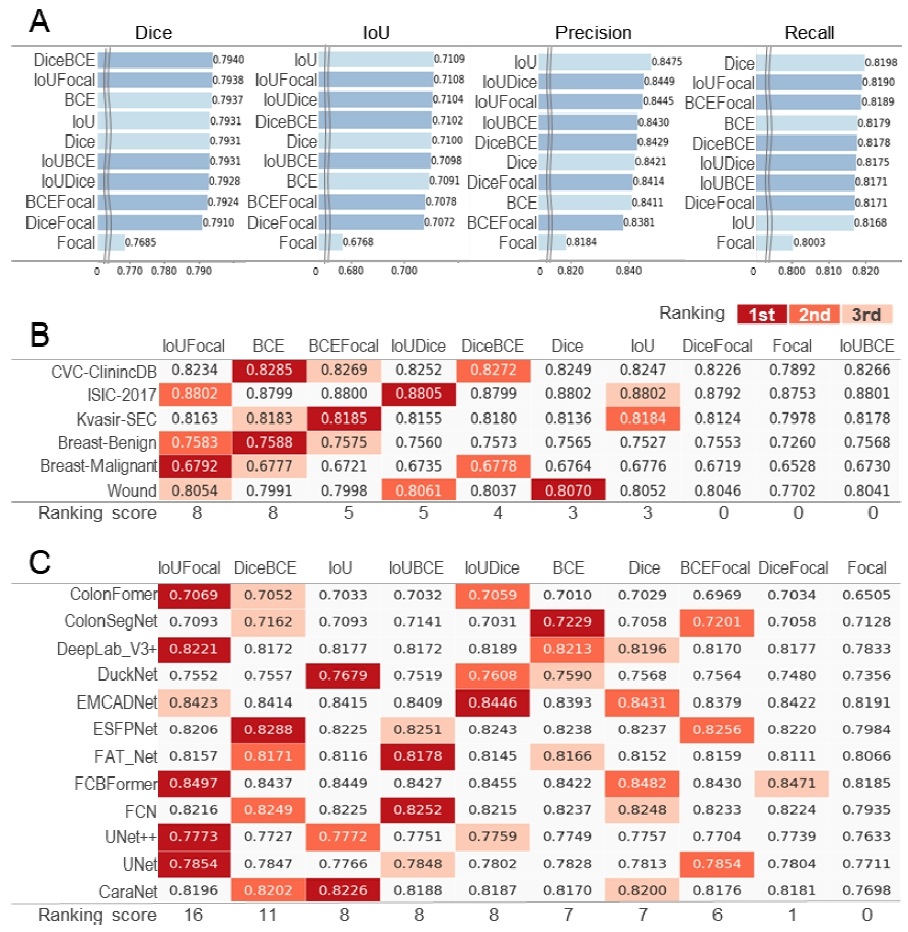
Comparison of accuracy performance of loss functions(A) Comparison of overall accuracy performance for 10 loss functions. Dark blue bars denote composite loss functions, while light blue bars denote simple loss functions. (B) Dice performance comparison by dataset. (C) Dice performance comparison by model. In (B) and (C), the intensity of red coloring highlights the ranks of 1st, 2nd, and 3rd within each dataset or model. The ranking score is calculated by assigning 3, 2, and 1 points for 1st, 2nd, and 3rd place, respectively, and then summing these points.
2. 데이터셋별, 모델별 성능 비교
데이터셋에 따라 손실 함수의 Dice 정확도 성능을 비교했을 때, 데이터셋 종류에 따라 최고 정확도의 손실 함수는 달라지는 경향을 보였다(그림 2B). 또, 딥러닝 모델에 따른 Dice 손실 함수 성능을 비교하였을 때, 딥러닝 모델에 따라 최적의 손실 함수가 달라지는 경향도 확인할 수 있었다(그림 2C). 우리는 각 데이터셋 또는 딥러닝 모델에 대해서 1, 2, 3등을 조사하여 각각 3, 2, 1점 점수를 부여하고 합산하여 종합 점수를 계산하였을 때, 종합 점수에서 IoUFocal이 6종의 데이터셋에 대해서 종합 8점(그림 2B)과 12종의 딥러닝 모델에 대해서 종합 16점(그림 2C)으로 가장 높은 점수를 획득함을 확인하였다. 전체 딥러닝 모델에 대한 점수 기준으로 우수한 성능의 손실 함수는 IoUFocal, DiceBCE, IoUDice, IoUBCE, IoU, BCE, Dice, BCEFocal, DiceFocal, Focal 순서였다.
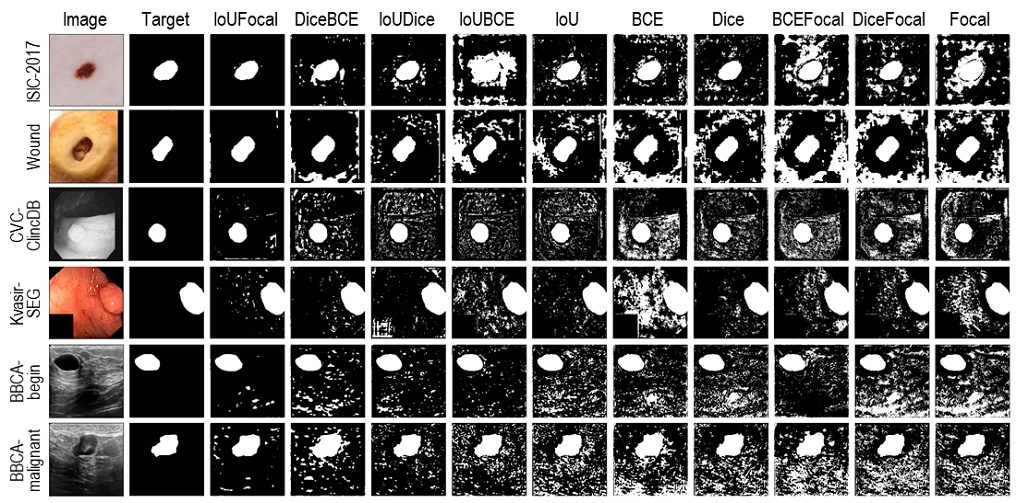
3. 사례 분석
우리는 손실 함수별로 예측 이미지의 사례를 시각화 나열하고 비교하여서 예측 정확도 성능이 차이 나는 원인을 분석하였다(그림 3). 일반적으로 딥러닝의 출력값에 대해서 Sigmoid 함수를 거친 후 0.5를 기준으로 병변 픽셀을 1, 일반 픽셀을 0으로 이진화하고 하였을 때, 시각화 결과에서 손실 함수별로 큰 차이를 확인할 수 없었다. 그러나 Sigmoid 함수를 거치지 않고, 출력의 평균값을 기준으로 병변 픽셀을 1, 일반 픽셀을 0으로 이진화하고, 이진화하여 시각화하였을 때 IoUFocal, DiceBCE 등 우리가 제시한 우수한 손실 함수들은 예측 결과가 병변과 일반 픽셀로 잘 이진화되는 경향을 보였으며, 종합 점수가 낮은 DiceFocal, Focal 등은 일반 배경에서 병변으로 예측하는 경향을 관찰하였다.
Ⅳ. 토의 및 결론
본 연구는 다양한 6개의 의료 영상 데이터셋과 12개의 딥러닝 모델, 그리고 10개의 손실 함수를 비교 분석하여 손실 함수가 딥러닝 모델 기반의 의료 영상의 병변 영역 분할 성능에 미치는 영향을 종합적으로 비교하였다. 실험 결과, 다양한 데이터나 모든 딥러닝 모델에서 절대적으로, 모두 일관되게 우수한 성능을 보이는 보편적으로 우수한 손실 함수는 나타나지 않았다. 데이터와 딥러닝 모델별로 특성이 다르기 때문에, 이러한 특성을 고려하여 적합한 손실 함수를 실험적으로 선택할 필요가 있음을 보여준다. 우리는 여러 종류의 병변 데이터셋과 딥러닝 모델 실험을 통해 IoUFocal 복합 손실이 여러 데이터셋과 딥러닝 모델에서 높은 종합 점수를 기록한 추천할 만한 손실 함수임을 조사하였다. 이 결과는 연구자가 본인 고유의 데이터셋이나 모델에 대해 최적의 손실 함수를 실험하여 조사할 수 없을 때, 우리의 실험 근거를 통해 적합한 손실 함수를 선택할 수 있도록 유의미한 지침을 제공한다. 사례 시각화 분석에서는 손실 함수가 어떤 점이 종합적 성능 정확도에 영향을 미칠 수 있는지 확인하였다. 그 결과는 좋은 손실 함수일수록 병변과 병변이 아닌 픽셀을 명확하게 구분한다는 것이다. 이러한 결과는 병변 이미지 영역 분할에 대한 향상된 손실 함수를 개발하는 데 활용될 수 있다. 또한, 복합 손실 함수의 사용이 개별 손실 함수보다 일반적으로 더 나은 성능을 나타낼 수 있다는 기존 연구 결과도 재확인하였다[5]. 우리의 연구 결과는 의료 영상 분할을 포함한 다양한 응용 분야에서 효과적인 기준 자료로 활용될 것으로 기대된다.
Acknowledgments
본 연구는 2024년 중소벤처기업부의 기술혁신개발사업의 지원에 의한 연구임 [RS-2024-00443396].
이 논문의 연구 결과 중 일부는 한국방송·미디어공학회 2024년 추계학술대회에서 발표한 바 있음.
References
-
X. Tang, “The role of artificial intelligence in medical imaging research,” BJR| open, Vol.2, No.1, p.20190031, 2019.
[https://doi.org/10.1259/bjro.20190031]

-
T. Wiltgen, et al., “LST-AI: A deep learning ensemble for accurate MS lesion segmentation,” NeuroImage: Clinical, Vol.42, p.103611, 2024.
[https://doi.org/10.1016/j.nicl.2024.103611]

-
R. Azad, E. K. Aghdam, et al., “Medical image segmentation review: The success of U-Net,” IEEE Transactions on Pattern Analysis and Machine Intelligence, Vol.46, No.12, pp.10076-10095, 2024.
[https://doi.org/10.1109/TPAMI.2024.3435571]

-
S. Jadon, “A survey of loss functions for semantic segmentation,” Proceeding of IEEE Conference on Computational Intelligence in Bioinformatics and Computational Biology (CIBCB), pp.1-7, 2020.
[https://doi.org/10.1109/CIBCB48159.2020.9277638]

-
J Ma, et al., “Loss odyssey in medical image segmentation,” Medical Image Analysis, Vol.71, p.102035, 2021.
[https://doi.org/10.1016/j.media.2021.102035]

-
J. Long, E. Shelhamer, and T. Darrell, “Fully convolutional networks for semantic segmentation,” Proceedings of the IEEE Conference on Computer Vision and Pattern Recognition, pp.3431-3440, 2015.
[https://doi.org/10.1109/CVPR.2015.7298965]

-
L. C. Chen, et al., “Encoder-decoder with atrous separable convolution for semantic image segmentation,” Proceedings of the European Conference on Computer Vision (ECCV), pp.801-818, 2018.
[https://doi.org/10.1007/978-3-030-01234-2_49]

-
O. Ronneberger, P. Fischer, and T. Brox, “U-Net: Convolutional Networks for Biomedical Image Segmentation,” Proceedings of Medical Image Computing and Computer-Assisted Intervention (MICCAI), pp.234-241, 2015.
[https://doi.org/10.1007/978-3-319-24574-4_28]

-
Z. Zhou, et al., “UNet++: A Nested U-Net Architecture for Medical Image Segmentation,” Proceeding of 4th International Workshop, DLMIA 2018, and 8th International Workshop, ML-CDS 2018, Held in Conjunction with MICCAI 2018, pp.3-11, 2018.
[https://doi.org/10.1007/978-3-030-00889-5_1]

-
D. Jha, et al., “Real-time polyp detection, localization and segmentation in colonoscopy using deep learning,” IEEE Access, Vol.9, pp.40496–40510, 2021.
[https://doi.org/10.1109/ACCESS.2021.3063716]

-
H. Wu, et al., “FAT-Net: Feature adaptive transformers for automated skin lesion segmentation,” Medical Image Analysis, Vol.76, p.102327, 2022.
[https://doi.org/10.1016/j.media.2021.102327]

-
E. Sanderson and B. J. Matuszewski, “FCN-transformer feature fusion for polyp segmentation,” Proceeding of Annual Conference on Medical Image Understanding and Analysis (MIUA), pp.892-907, 2022.
[https://doi.org/10.1007/978-3-031-12053-4_65]

-
N. T. Duc, et al., “ColonFormer: An efficient transformer-based method for colon polyp segmentation,” IEEE Access, Vol.10, pp.80575-80586, 2022.
[https://doi.org/10.1109/ACCESS.2022.3195241]

-
A. Lou, S. Guan, and M. H. Loew, “CaraNet: Context Axial Reverse Attention Network for segmentation of small medical objects,” Journal of Medical Imaging, Vol.10, No.1, p.014005, 2023.
[https://doi.org/10.1117/1.JMI.10.1.014005]

-
Q. Chang, et al., “ESFPNet: Efficient deep learning architecture for real-time lesion segmentation in autofluorescence bronchoscopic video,” Proceeding of Medical Imaging 2023: Biomedical Applications in Molecular, Structural, and Functional Imaging, p.1246803, 2023.
[https://doi.org/10.1117/12.2647897]

-
R. G. Dumitru, D. Peteleaza, and C. Craciun, “Using DUCK-Net for polyp image segmentation,” Scientific Reports, Vol.13, No.1, p.9803, 2023.
[https://doi.org/10.1038/s41598-023-36940-5]

-
M. M. Rahman, M. Munir, and R. Marculescu, “EMCAD: Efficient multi-scale convolutional attention decoding for medical image segmentation,” Proceedings of the IEEE/CVF Conference on Computer Vision and Pattern Recognition, pp.11769-11779, 2024.
[https://doi.org/10.1109/CVPR52733.2024.01118]


- 2021년 ~ 현재 : 수원대학교 데이터과학부 학사
- ORCID : https://orcid.org/0009-0004-8331-3033
- Homepage : https://sou414.tistory.com
- 주관심분야 : Medical image AI, Medical Image Processing

- 2019년 : 서울대학교(Seoul National University) 컴퓨터공학 박사
- 2019년 ~ 2020 : 서울대학교(Seoul National University) 박사 후 연구원
- 2020년 ~ 현재 : 수원대학교 데이터과학부 조교수
- 2021년 ~ 현재 : 수원대학교 DS&ML 센터장
- ORCID : https://orcid.org/0000-0002-5941-9784
- Homepage : https://hongryulahn.github.io/
- 주관심분야 : Bio and health informatics, AI appication